진핵생물 분류를 위한 EUKARYOME database (2024)
EUKARYOME (ver 1.8)
- 논문 게재: 2024.05.23
- Metazoans, protists, fungi와 plants를 모두 포함하는 all eukaryotes에 대한 데이터 베이스
- General FASTA (for DADA2), QIIME, Mothur, BLAST 전용 DB 제공
- 18S의 Long, SSU, LSU, ITS 총 네 가지 데이터 제공
기존 데이터 베이스의 한계
특정 영역만 다룬다 & 업데이트가 느림(PR2, SILVA/ 2020)
- UNITE: ITS
- PR2 database: SSU
- SILVA: SSU & LSU
=> SSU, LSU, ITS를 모두 담은 DB는 없을까?
제작방법
- Amplicon(SILVA v138.1 + PR2 v4.14.1 + UNITE v 9.0) + Full-length(INSDc 16/04/2023) + PacBio HiFi + Oxford nanopore consensus
- Multiple sequence alignments using MAFFT v7 with standard options
사용법 for ITS
DADA2
1. https://eukaryome.org/blast/ 접속
2. General_EUK_ITS 다운로드 (1,069,617 sequences) 이후 압축 풀기
3. FASTA 파일 형식 수정
- DADA2에 적합하게 설계되어 있지 않다.
- linux상에서 아래 스크립트를 통해 수정(SILVA형식으로 변환)
`sed -e 's/^>[^;]*;/>/' General_EUK_ITS_v1.8.fasta > General_EUK_ITS_v1.8_modi.fasta`
`sed -e '/^>/ s/$/;/' General_EUK_ITS_v1.8_modi.fasta > General_EUK_ITS_v1.8_modi2.fasta`
4. DADA2에서 매칭
EUKv1.8 <- "/data/Reference/ITS/DADA2/EUKAYOME/ver1.8/General_EUK_ITS_v1.8_modi2.fasta"
taxa.EUK.QC30.its2 <- dada2::assignTaxonomy(ASVs, EUKv1.8, multithread = TRUE, tryRC = TRUE)
write.table(as.data.frame(taxa.EUK.QC30.its2), "./output/3.assignment/QC30_tax_forward_EUKver1.8.txt", quote = F)
QIIME2
1. https://eukaryome.org/blast/ 접속
2. Linux환경에서
wget https://sisu.ut.ee/wp-content/uploads/sites/643/QIIME2_EUK_ITS_v1.8.zip
qiime tools import --type 'FeatureData[Taxonomy]' \
--input-path QIIME2_EUK_ITS_v1.8.tsv \
--output-path QIIME2_EUK_ITS_v1.8_taxonomy.qza
qiime tools import --type 'FeatureData[Sequence]' \
--input-path QIIME2_EUK_ITS_v1.8.fasta \
--output-path QIIME2_EUK_ITS_v1.8_fasta.qza
qiime feature-classifier fit-classifier-naive-bayes \
--i-reference-reads QIIME2_EUK_ITS_v1.8_fasta.qza \
--i-reference-taxonomy QIIME2_EUK_ITS_v1.8_taxonomy.qza \
--o-classifier QIIME2_EUK_ITS_v1.8_classifier.qza
## 사용
qiime feature-classifier classify-sklearn \
--i-classifier classifier.qza \
--i-reads your_sequences.qza \
--o-classification taxonomy.qza
에러 및 해결
| 에러 🚨
- DADA2에서 UNITE v8.2, UNITEv10.0, THF 1.6.1, EUKAYOMEv1.8을 비교하고자 약 1,600개의 ASV를 Assignment
- UNITE v8.2 및 v10.0 데이터베이스를 사용할 때, 모든 시퀀스가 균계(Kingdom) 수준에서 Fungi (1,607 ASVs)
- EUKAYOMEv1.8 에서는 Kingdom-level에서 약 1,100개의 NA, 또한 Fungi로 매칭된 Kingdom은 오직 250개
| 해결
1. 데이터가 너무 커서 그런가?
- DB size: SILVA> EUKAYOME > RDP > UNITE
=> 10% 만 추출후 매치 -> 일부 NA가 뜨지만 전보다는 나아짐
2. CPU 과부화 때문인가?
- thread 의 개수를 제한을 두고 수행
=> 차이 없음
3. EUKARYOME의 서열이 기존 UNITE서열과 차이 나는가?
- M.restricta서열만 추출, EUKARYOMEv1.8 (104개)/ UNITEv10(4개)
=> 계통수 구축 시 큰 차이 ㄴㄴ
4. RDP classifier문제라고 가정
- RDP classifier란?

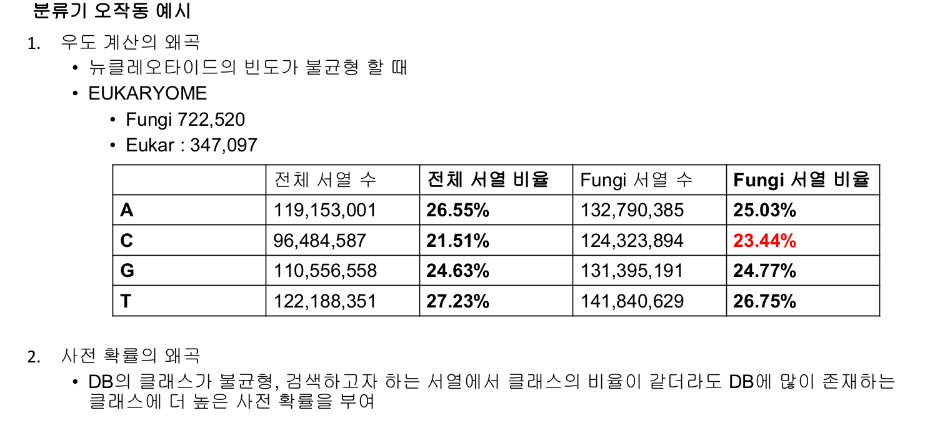
- 그렇다면 노이즈 때문인가?

=> 맞았다.
그러므로 Fungi 만 추출 후 사용하는 것을 추천드립니다!