[Microbiome] 온라인으로 간편하게 amlicon, shotgun 분석하기 (Nephele)
오늘 발견한 Microbiome 분석 사이트를 소개한다. 이는 파일만 업로드하면 원하는 분석 플랫폼(dada2, qiime2, muthur)으로 분석한 결과를 보내주는 사이트이다. NIH의 연구진이 개발하였다.
이 분석으로 논문 데이터를 분석하는 것도 가능하지만, 처음 분석에 접하는 사람들을 대상으로 교육할 때, 도움이 될 것 같아 글을 적어본다.
| 주소
- https://nephele.niaid.nih.gov/index
Nephele
Explore Run additional analysis and visualizations
nephele.niaid.nih.gov
| 분석 방법
1. 회원 가입
2. 원하는 분석 선택하기
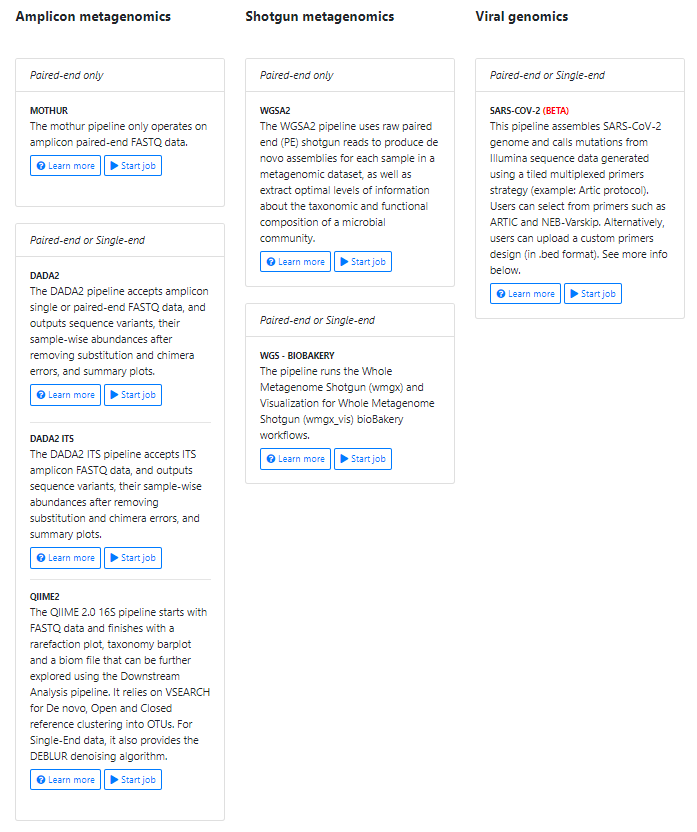
여기서 dada2_ITS를 선택해 보겠다.
이후 시퀀싱 된 서열의 형식을 선택해 준다.

3. 파일 업로드
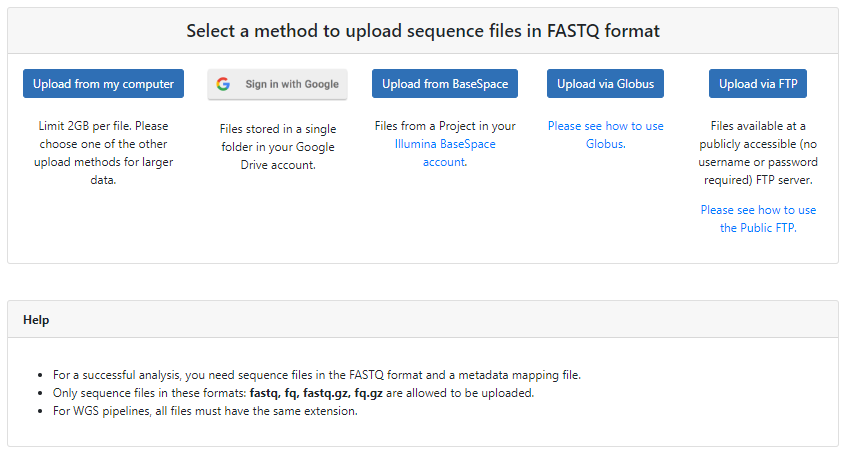
업로드 형식을 지정해 준다.
컴퓨터 안에 있는 파일을 사용할 것이니, Upload from my computer를 클릭(2GB 제한)

Add files를 클릭 한 뒤, 원하는 파일을 선택합니다. 이후, Start upload를 눌러, Nephele서버에 업로드한다.
4. metadata 첨부

샘플의 정보를 추가해 준다. 양식은 Download mapping file template 통해 양식을 파악할 수 있다. 이는 qiime2의 양식과 유사하다.
5. 분석 옵션 지정
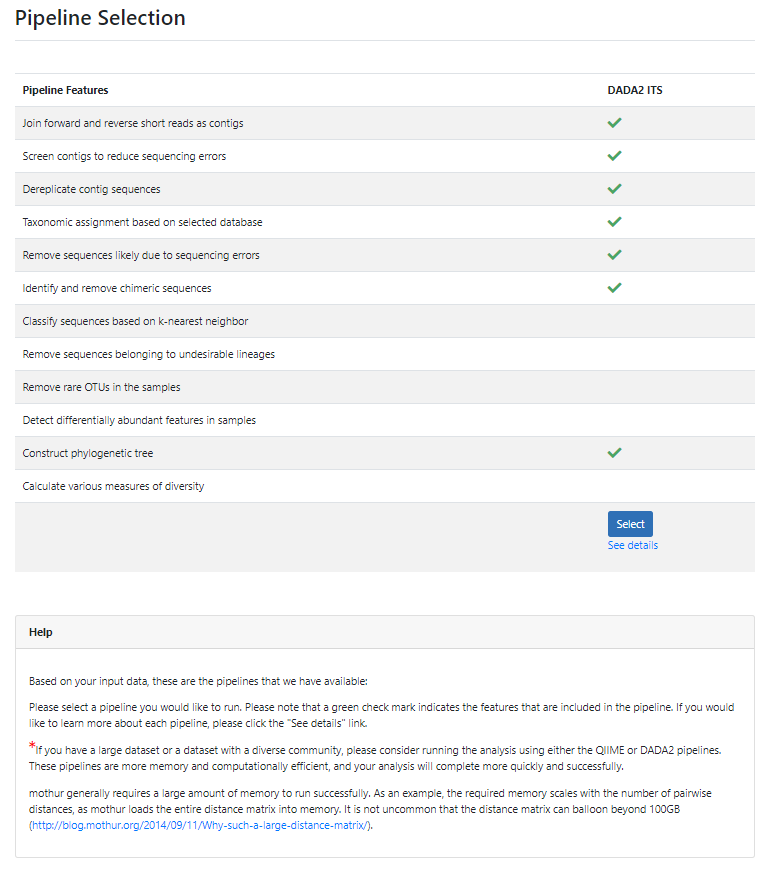
원하는 분석 옵션을 지정한다. 일단 수정하지 않고 기본 값으로 분석을 진행하였다.
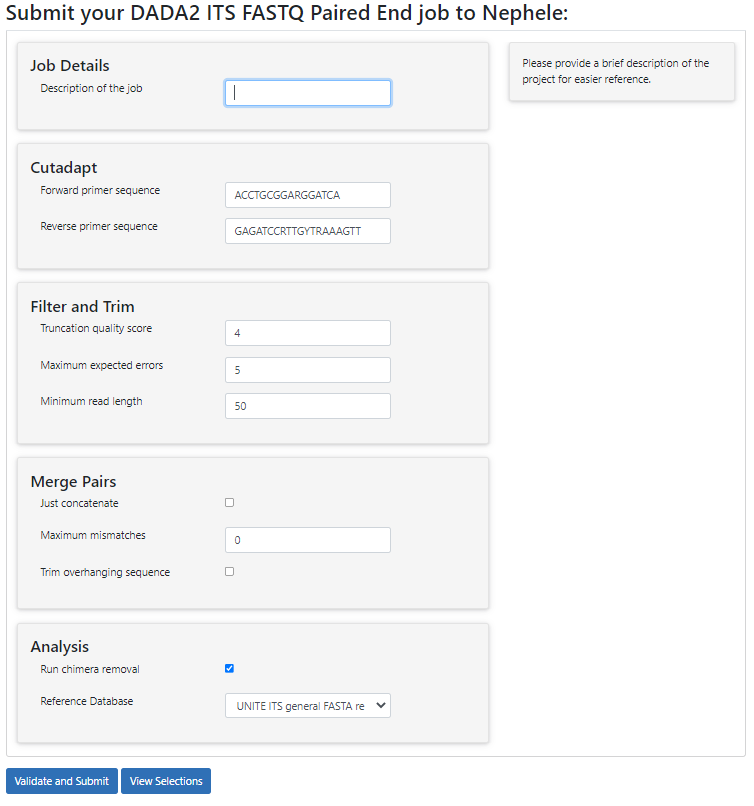
조금 더 디테일한 분석 옵션을 설정할 수 있다. 각 단계의 설명은 이곳을 참고.
이것도 수정하지 않고 기본 값으로 설정하였다.
| 결과물

제출이 완료되었으며, 회원가입 시 사용했던 이메일로 결과가 전송된다. 이는 데이터 크기에 따라 소요되는 시간이 다르다.
도착한 첫 번째 메일은 분석한 값의 옵션이며, 두 번째 메일이 실제 샘플의 분석 결과이다.

받은 메일에서 결과 파일을 다운로드하여보자. 결과 파일은 아래의 값들이 있다.
a. dada2 분석 결과

b. QC plot

c. OTU and Taxonomy table

d. Diversity


e. Tree 파일
f. heat map
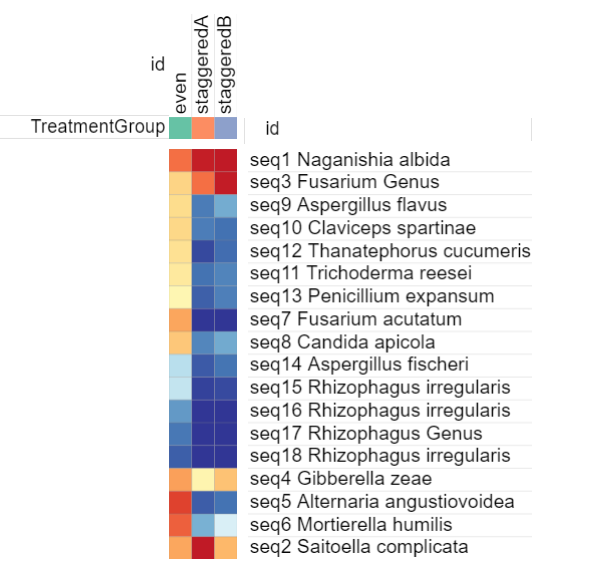
g. 중간 결과물 파일
h. 완성된 ASV 서열 파일