[QIIME2] FungiDB THF database를 QIIME버전으로 바꾸어 보자

Fungi database에는 UNITE가 가장 대표적으로 사용됩니다. 그러나 Targeted Host-associated Fungi ITS Database(THF)도 유명한 데이터베이스 중 하나입니다. 이를 dada2 버전으로 바꾼 다음에 사용해 봅시다.

1. 파일 다운로드
가장 최신 버전인 1.6.1을 다운로드하여 보겠습니다.
- THFv1.6.1
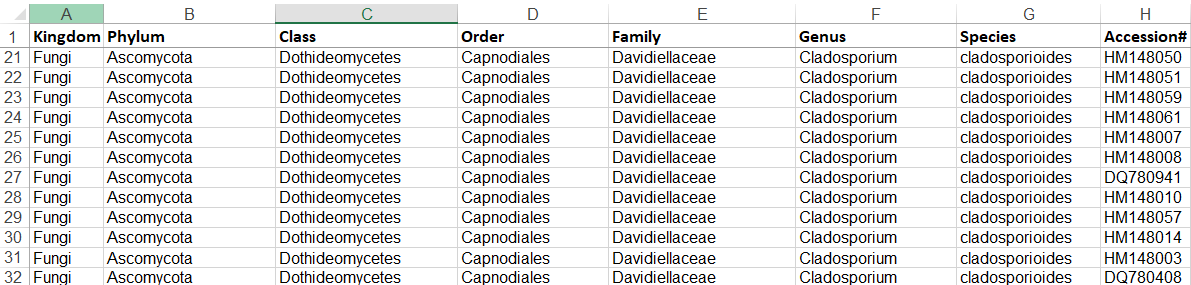
- FASTA sequencesv1.6.1
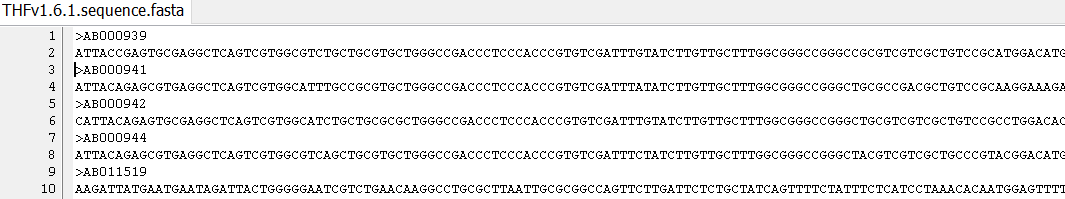
위 파일은 QIIME2에 사용하기 좋은 형식으로 나누어져 있습니다. 즉 taxonomy annotation정보와 sequence정보가 따로 저장되어 있습니다.
이때 Accession 번호를 통해서, 각 서열에 매치된 데이터베이스의 정보를 파악할 수 있습니다.
2. Taxonomy 형식 바꾸기
그러나 QIIME2 형식은 아래처럼, FeatureID와 taxon으로 구성된 형식( HeaderlessTSVTaxonomyFormat )을 많이 사용합니다.
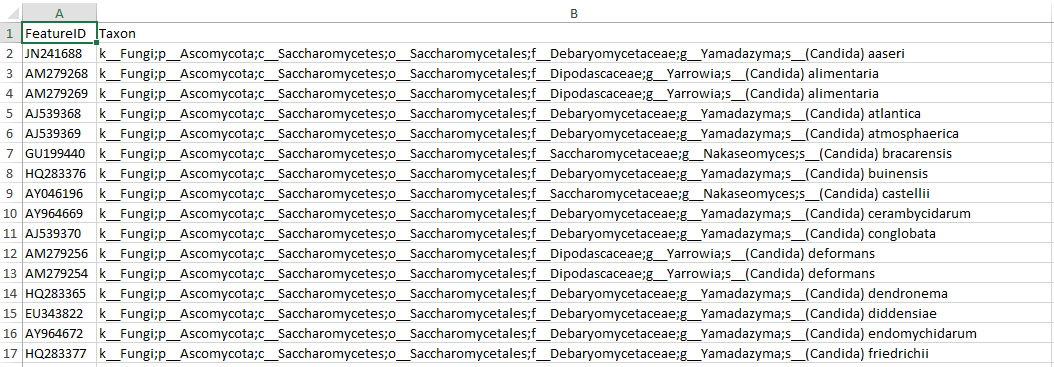
위 이미지처럼 taxonomy 데이터를 수정해 준 후, UTF-8 형식의 txt 혹은 tsv로 저장해 줍니다.
3. Linux에서 qiime2 artifact으로 변환하기
## 서열 데이터
qiime tools import --type 'FeatureData[Sequence]' \
--input-path THFv1.6.1.sequence.fasta \
--output-path THFv1.6.1.sequence.qza
#Imported THFv1.6.1.sequence.fasta as DNASequencesDirectoryFormat to THFv1.6.1.sequence.qza
## 계통 정보 데이터
qiime tools import --type 'FeatureData[Taxonomy]' \
--input-format HeaderlessTSVTaxonomyFormat \
--input-path THFv1.6.1_for_qiime2.txt \
--output-path THFv1.6.1_for_qiime2.qza
#Imported THFv1.6.1_for_qiime2.txt as HeaderlessTSVTaxonomyFormat to THFv1.6.1_for_qiime2.qza
## qiime2 형식의 분류기 만들기
qiime feature-classifier fit-classifier-naive-bayes \
--i-reference-reads THFv1.6.1.sequence.qza \
--i-reference-taxonomy THFv1.6.1_for_qiime2.qza \
--o-classifier THFv1.6.1_classifier.qza
#Saved TaxonomicClassifier to: THFv1.6.1_classifier.qza
이렇게 외부 데이터를 qiime2형식의 database로 간단하게 변환이 가능하다.
[DADA2] FungiDB THF database를 dada2 버전으로 바꾸어 보자
Fungi database에는 UNITE가 가장 대표적으로 사용됩니다. 그러나 Targeted Host-associated Fungi ITS Database(THF)도 유명한 데이터베이스 중 하나입니다. 이를 dada2 버전으로 바꾼 다음에 사용해 봅시다. 1.
bio-kcs.tistory.com