[likert] 항생제 내성률을 likert plot으로 시각화하기

Likert plot이란?
Likert plot은 흔히 설문조사라고 불리는 리커트 척도(likert scale) 조사에 사용되는 시각화 방법입니다.
리커트 척도는 문장을 제시하고, 그에 대한 동의/비동의를 평가하는 방식을 뜻한다. 주로 3~5가지의 문항을 사용한다고 합니다.
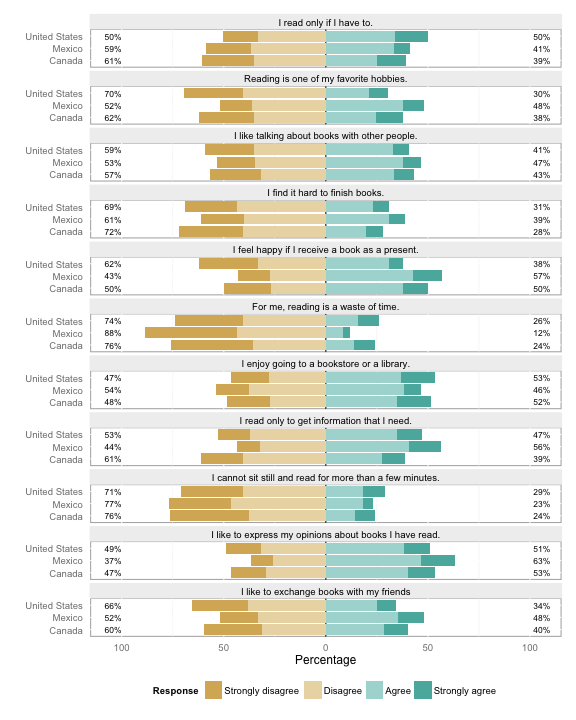
R의 likert plot은 원데이터를 분포로 바꾸지 않아도, 자동적으로 요약 및 시각화를 해주는 도구입니다.
항생제 내성결과 또한 분포에 따라 데이터를 정렬하는 것으로, likert plot을 사용하기 좋은 데이터입니다.
항생제 내성 결과
항생제 내성은 3가지로 나눌 수 있습니다.
- R (Resistant): high likelihood of therapeutic failure.
- S (Susceptible) high likelihood of therapeutic success
- I (Susceptible) high likelihood of therapeutic success, but only when exposure to an antimicrobial agent is increased
항생제 보고서의 예시는 아래와 같습니다.
| patient | date | test_no | specimen | mo | PEN | AMC | CIP |
| 1 | 2019-03-08 | 100 | blood | esccol | R | I | S |
| 1 | 2019-03-09 | 101 | blood | esccol | R | I | S |
| 2 | 2019-03-08 | 102 | blood | staaur | R | S | - |
| 3 | 2019-03-08 | 103 | urine | pseaer | R | R | R |
mo는 배양된 균(약어로 표시)과 각 항생제의 약어에 따른 내성결과가 적혀있습니다.
또한, 항생제 내성분석에서는 범주형 변수의 통계 분석 방법인 Chi-square Test, Fisher's Exact Test 가 주로 사용됩니다. Chi-squere의 조건을 맞추기 어려우니, Fisher's Exact Test를
항생제 내성 결과 likert plot으로 시각화하기
자, 예제데이터와 함께, likert package를 사용해서 시각화해 봅시다!
library(likert)
library(ggplot2)
library(dplyr)
# 데이터 생성
set.seed(123) # 재현성을 위해 시드 설정
example_data <- data.frame(
No = seq(1:30),
gender = sample(c("F", "M"), 30, replace = TRUE),
Age = sample(20:70, 30, replace = TRUE),
Group = rep("Disease", 15) %>% c(rep("Healthy", 15)),
mo = paste0("Species_", seq(1:30)),
CIP = c(sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.6, 0.3, 0.1)), sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.2, 0.6, 0.2))),
GEN = c(sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.6, 0.3, 0.1)), sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.2, 0.6, 0.2))),
SXT = c(sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.6, 0.3, 0.1)), sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.2, 0.6, 0.2))),
QDA = c(sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.6, 0.3, 0.1)), sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.2, 0.6, 0.2))),
LZD = c(sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.6, 0.3, 0.1)), sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.2, 0.6, 0.2))),
TEC = c(sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.6, 0.3, 0.1)), sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.2, 0.6, 0.2))),
VAN = c(sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.6, 0.3, 0.1)), sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.2, 0.6, 0.2))),
PEN = c(sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.6, 0.3, 0.1)), sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.2, 0.6, 0.2))),
CLI = c(sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.6, 0.3, 0.1)), sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.2, 0.6, 0.2))),
ERY = c(sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.6, 0.3, 0.1)), sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.2, 0.6, 0.2))),
FUS = c(sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.6, 0.3, 0.1)), sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.2, 0.6, 0.2))),
HAB = c(sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.6, 0.3, 0.1)), sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.2, 0.6, 0.2))),
MUP = c(sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.6, 0.3, 0.1)), sample(c("R", "S", "I"), 15, replace = TRUE, prob = c(0.2, 0.6, 0.2)))
)
example_data질환군과 정상군을 나누어 예제데이터를 만들어보았습니다. 항생제의 이름은 약어로 표시하였습니다.
또한 질환군에서 R 값이 더 많은 분포를 가지도록 확률을 조정하였습니다.
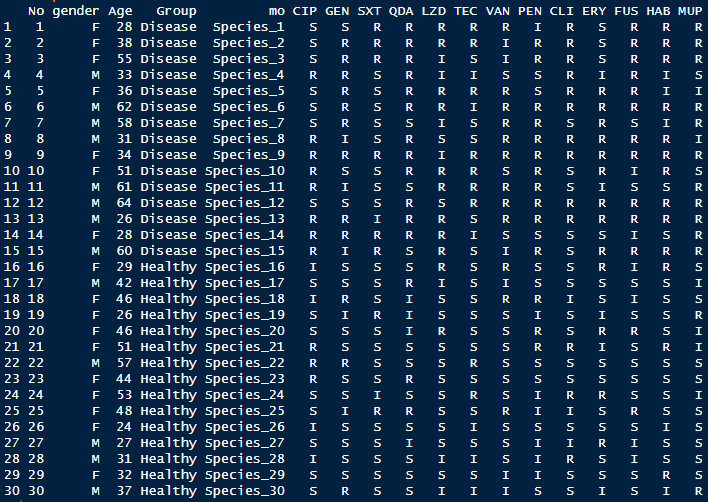
질환군의 데이터만 추출한 후 항생제 내성률을 관찰해 봅시다.
여기서 중요한 포인트는 1) 데이터가 factor 형식이어야 하며, 2) likert()의 input 데이터는 data.frame형태여야 합니다.
추가로 NA 값은 있어도 상관없습니다.
example_data_di <- example_data[example_data$Group %in% "Disease", ]
disease <- lapply(example_data_di[ ,9:18] , factor, levels= c("S", "I", "R")) %>%
as.data.frame()
disease.li <- likert(items = disease)
print(disease.li)
# Item S I R
# 1 QDA 26.666667 13.333333 60.00000
# 2 LZD 40.000000 13.333333 46.66667
# 3 TEC 46.666667 6.666667 46.66667
# 4 VAN 20.000000 6.666667 73.33333
# 5 PEN 26.666667 6.666667 66.66667
# 6 CLI 6.666667 6.666667 86.66667
# 7 ERY 33.333333 13.333333 53.33333
# 8 FUS 20.000000 6.666667 73.33333
# 9 HAB 33.333333 26.666667 40.00000
# 10 MUP 53.333333 6.666667 40.00000
plot(likert(disease))
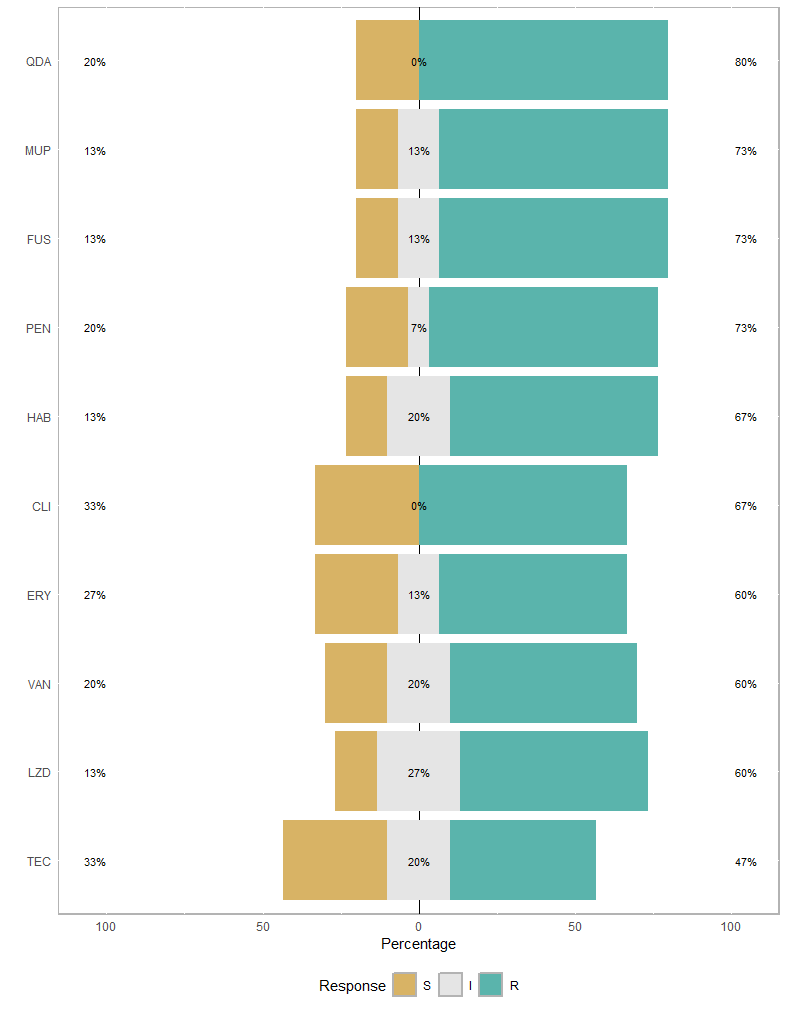
내성률이 높은 것을 확인할 수 있습니다.
정상대조군의 내성률을 확인해 봅시다!
example_data_he <- example_data[example_data$Group %in% "Healthy", ]
healthy <- lapply(example_data_he[ ,9:18] , factor, levels= c("S", "I", "R")) %>%
as.data.frame()
healthy.li <- likert(items = healthy)
plot(healthy.li)
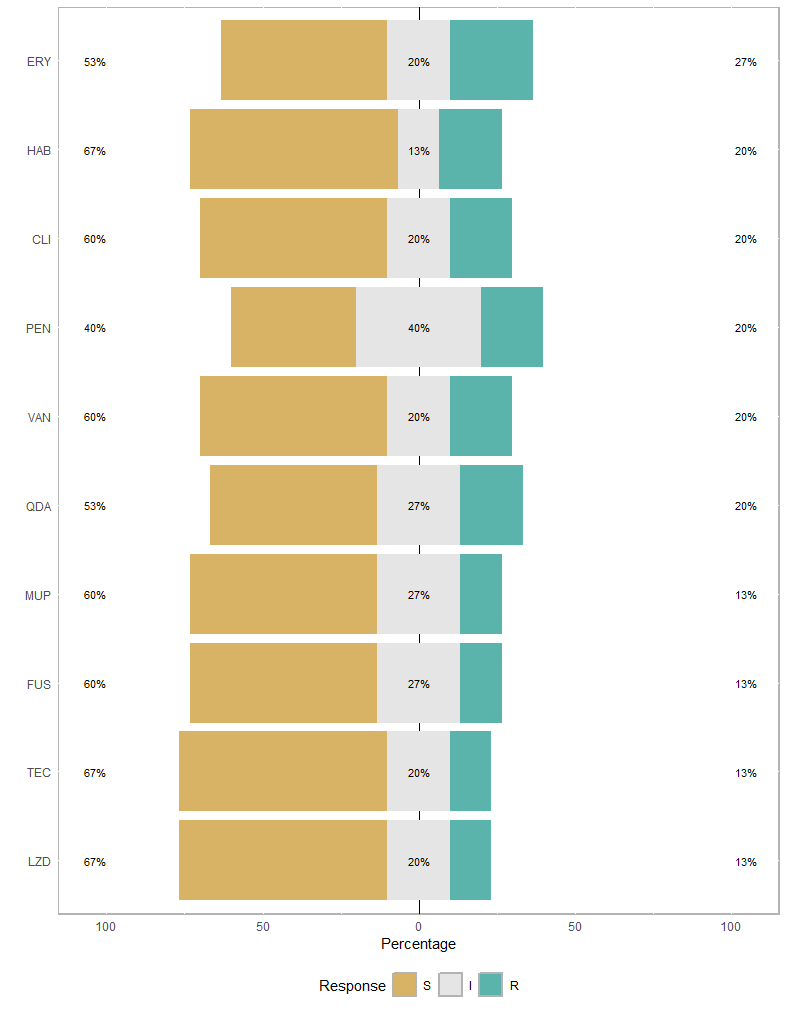
반대로 sensitive 한 비율이 더 높은 것을 볼 수 있습니다.


이렇게 같이 보면 항생제에 따른 내성률의 확연한 차이를 관찰할 수 있습니다.
축을 바꾸고 싶다면, center옵션을 사용하실 수 있습니다.
plot(healthy.li, center=1.5)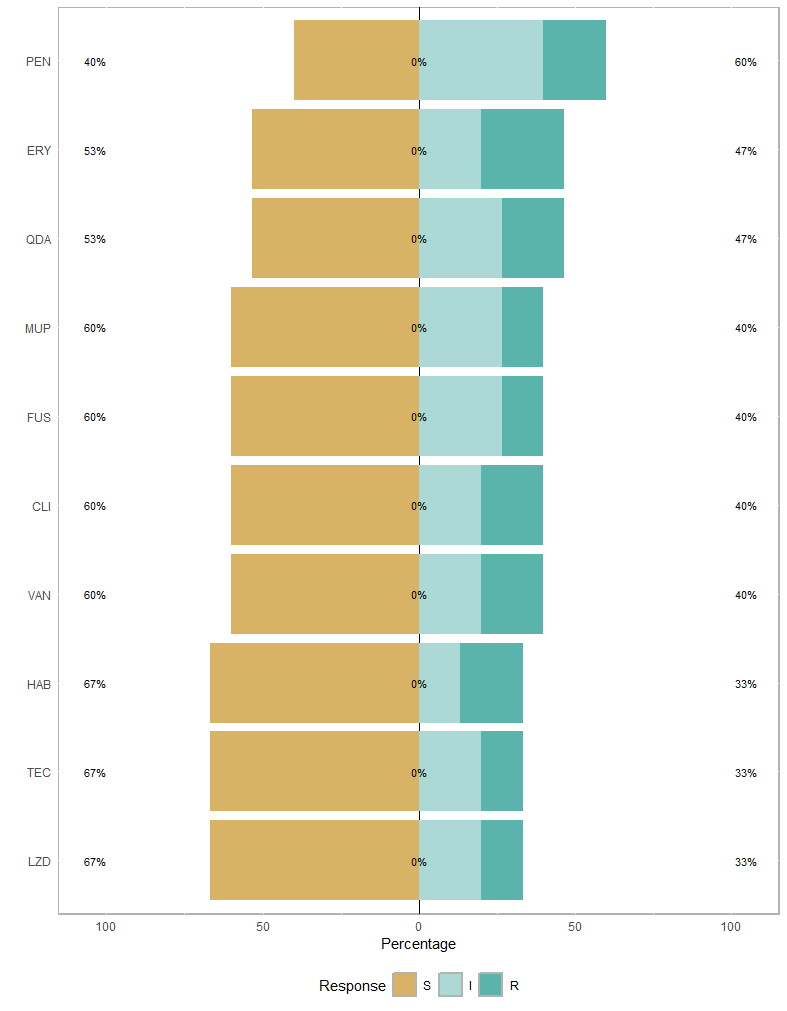
plot(healthy.li, center=2.5)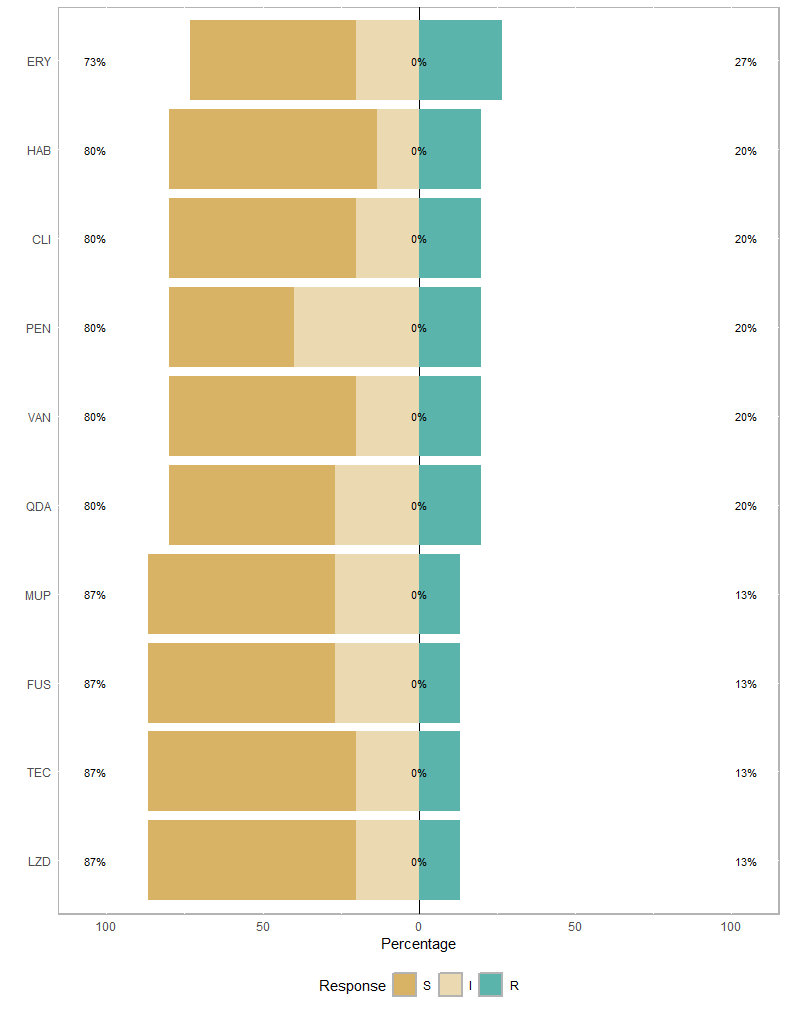
알아두면 좋은 패키지
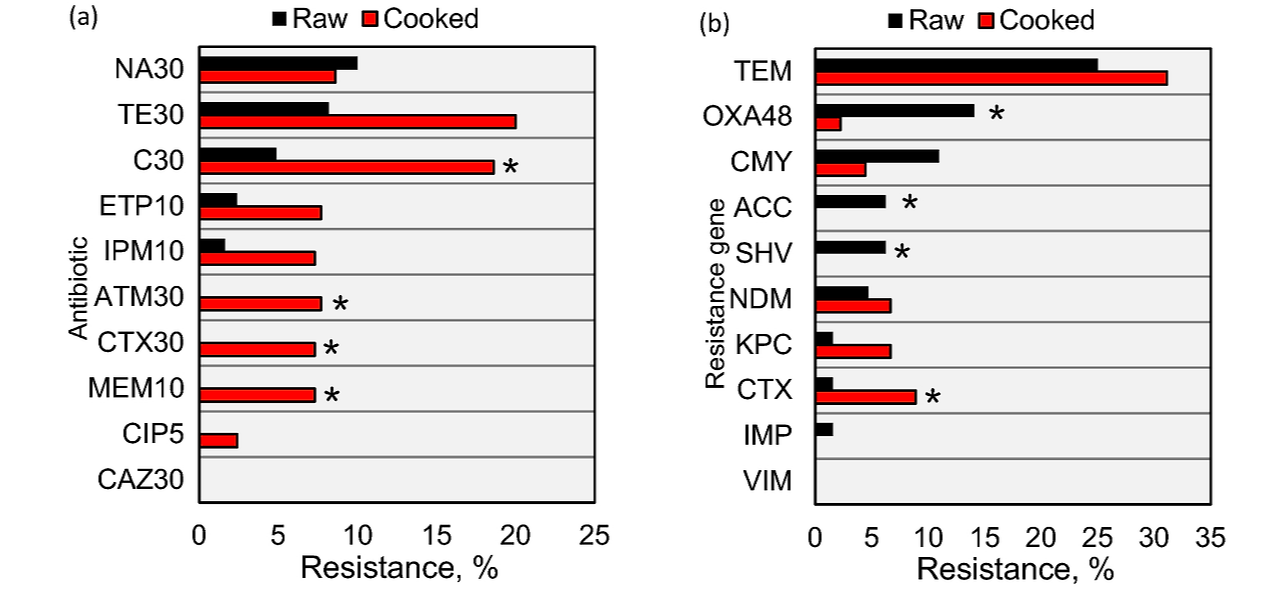
- 항생제 내성 결과를 분석하고 시각화하는 전문 패키지인 AMP (https://msberends.github.io/AMR/index.html) 이 따로 존재합니다. 이 패키지는 여러 기능을 담고 있는데, 예를 들어 Propionibacterium acne를 Cutibacterium acne로 자동적으로 바꾸어 주고, 나라마다 일부 차이가 있는 항생제 약어를 통합시켜주기도 합니다(한국은 포함되어 있지 않음ㅜ).
물론 아래와 같이 내성률을 시각화하거나, 각 균에 따른 항생제의 영향을 biplot으로 보여주기도 합니다.
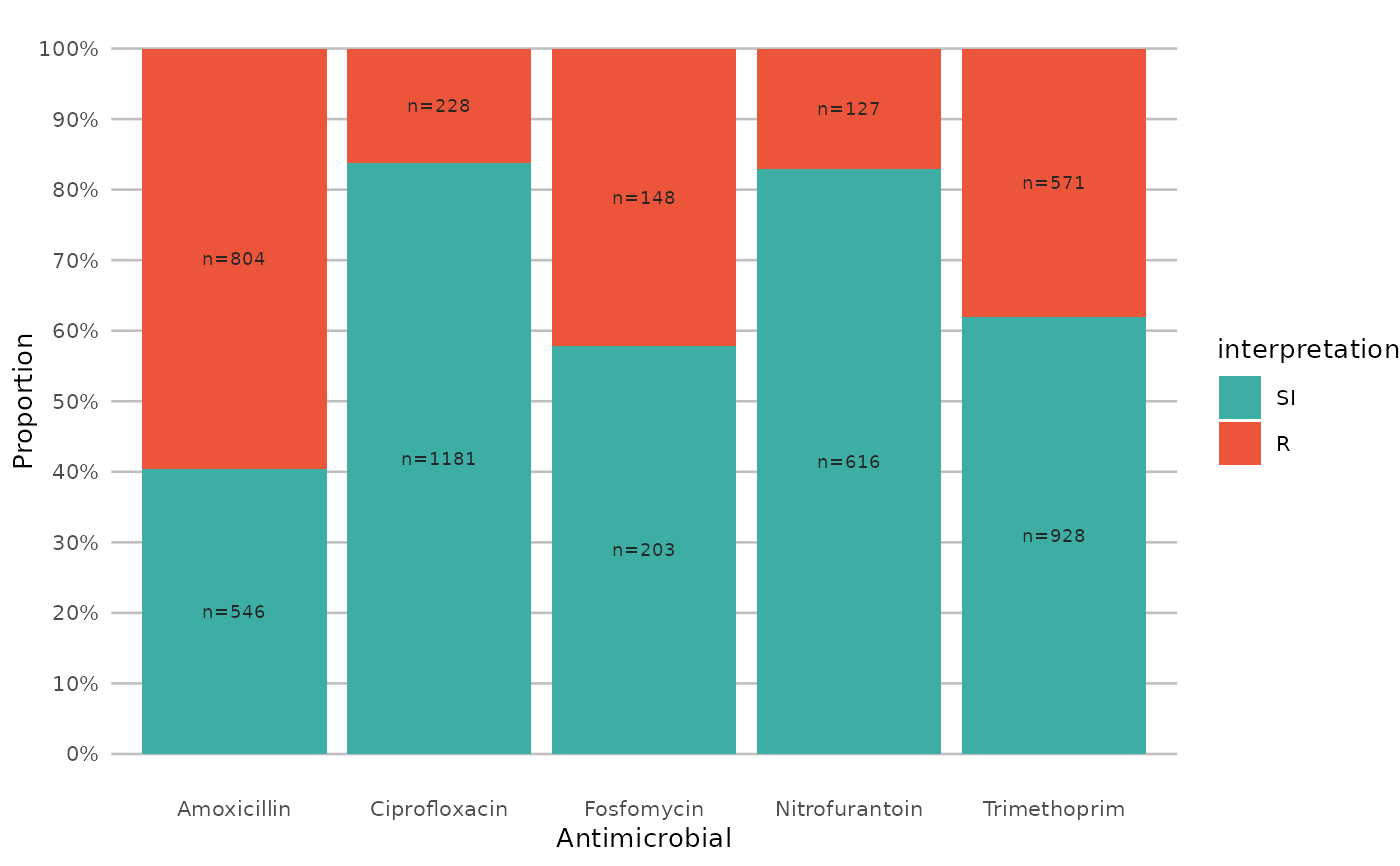

input 데이터 형식을 맞추어야 하는 번거로움이 있지만, 항생제 내성데이터를 전문적으로 다루신다면 편리한 도구가 될 것입니다.
참고
- Berends, M. S., Luz, C. F., Friedrich, A. W., Sinha, B. N. M., Albers, C. J., & Glasner, C. (2022). AMR: An R Package for Working with Antimicrobial Resistance Data. Journal of Statistical Software, 104(3), 1–31. https://doi.org/10.18637/jss.v104.i03
