검색을 하다가 여러 카테고리를 가진 해외 커뮤니티를 발견하였다. 레딧은 여러 주제별로 다양한 이야기를 토론하는 사이트인듯하다. 여기 bioinfomatics 레딧에 유용한 정보가 많이 올라오니 구독을 추천한다.
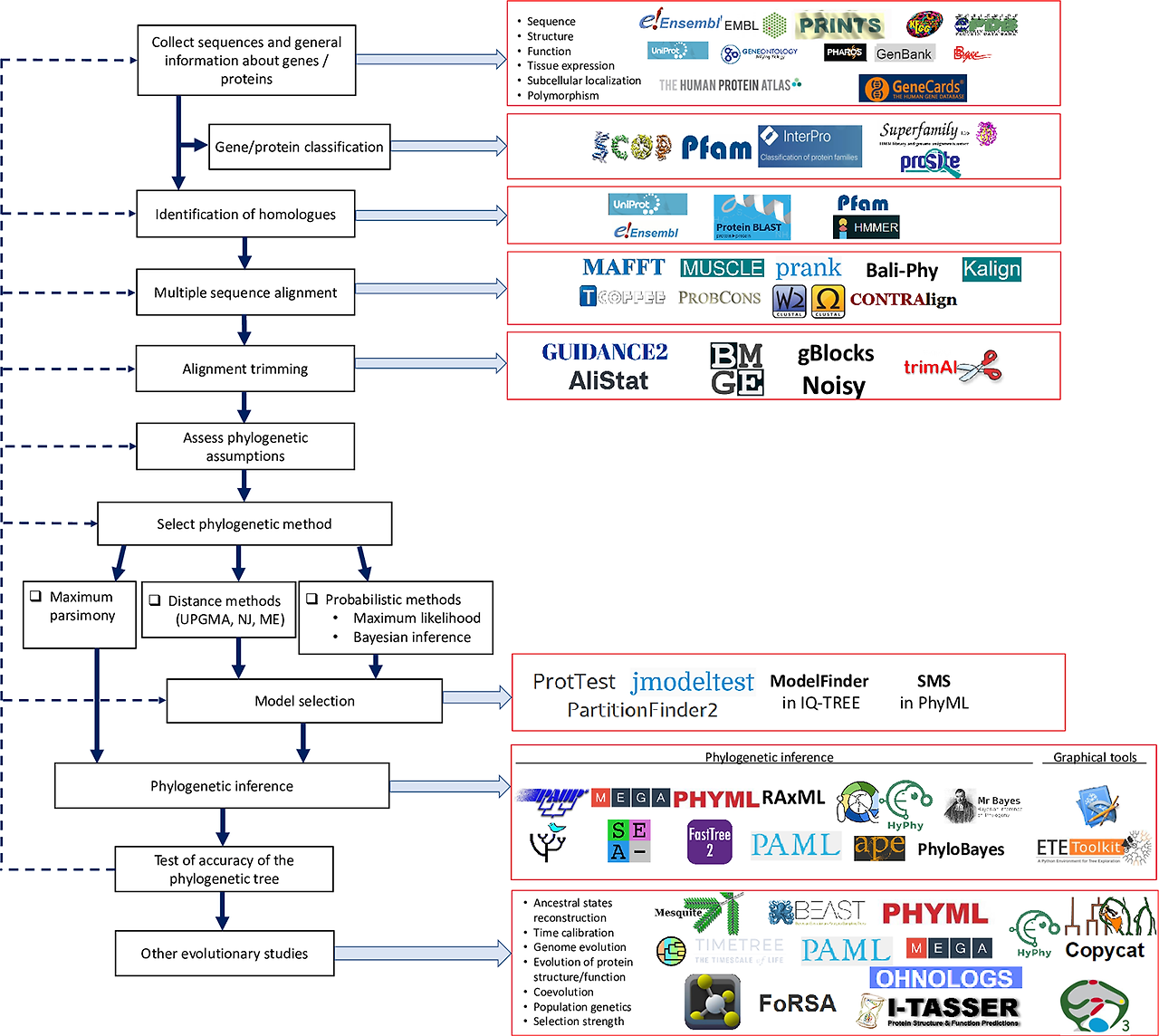
현재 Fungi의 분석 pipeline을 구축 중인데, 계통수에 관한 글을 가져왔다.
1. https://www.reddit.com/r/bioinformatics/comments/13w13ra/building_phylogenetic_trees/
| 정리하자면..
- dataset이 작다면 DECIPHER로 정렬 후 phangorn으로 계통수 제작
+) 근데 정렬은 이왕이면 R보다는 muscle에서 multiple sequence alignment(MSA)로 수행해라
- dataset이 크다면, Raxml, iqtree로 계통수 제작
| Raxml vs iqtree는?
- Raxml은 RAM사용이 효율적이지만, iqtree가 좀 더 빠르다는 의견이 있다(레딧)
- 논문 "An investigation of irreproducibility in maximum likelihood phylogenetic inference, 2020"에서는 아무리 같은 데이터라도 데이터 크기가 아닌 CPU에 따라서 9~18%는 재현하기 어렵다고 한다. 또한 Raxml보다 iqtree가 재현성이 더 높음. 그러나 multithreading 시에 iqtree는 거의 재현 불가능.. 최대한의 재현성을 보장하기 위해서는 alighment 알고리즘, 프로그램 이름, seed 값, 트리 검색 수, 대체 모델, 프로세서 유형 및 thread 수를 의무적으로 보고해야 한다.
참고
- https://evomics.org/wp-content/uploads/2024/01/Krumlov2024_raxng.pdf
- https://www.biorxiv.org/content/10.1101/2022.10.31.514545v1.full.pdf
- https://cme.h-its.org/exelixis/pubs/masterJulia.pdf
- https://www.biostars.org/p/9505307/
- https://genoglobe.com/kribb/bioinfo/%EA%B3%84%ED%86%B5%EC%88%98_%EC%9E%91%EC%84%B1%ED%95%98%EA%B8%B0
-- 위 블로그의 추천글 1: https://cran.r-project.org/web/packages/phytools/phytools.pdf
-- 2: http://www.phytools.org/Cordoba2017/ex/2/Intro-to-phylogenies.html
- https://github.com/benjjneb/dada2/issues/88
- https://www.reddit.com/r/bioinformatics/comments/13w13ra/building_phylogenetic_trees/
- https://github.com/amkozlov/raxml-ng

2019년도 글이지만, 계통분류학 분야는 이제 질문글을 올릴 때 컴퓨터의 사양을 같이 올리는 것이 주류인 듯하다.
아무래도 자유로운 커뮤니티다 보니, 학술 질문 형식보다는 자유롭게 물어보는 편이다. 근데 이 글은 너무 자유롭게 써서 계통수 제작 도구보다 말투에 대한 반응이 ↑가 더 많다🤣🤣
