수정: 2024.12.11

일단 다른 연구에 도움이 되고자, 기존 HMP 데이터 베이스를 재분석하는 일을 하였다.
교수님이 짬 날 때만 하라고 하셨지만, 짬이 많이 나서 빠르게 해치웠다.
위 프로젝트의 목표는 기존 데이터셋에 나타나지 않은 A라는 균이 우리 연구실 샘플에서 많이 나타나는데,
HMP 데이터에서 이 A가 과소평가된 게 아닐까? 하는 의구심으로 시작되었다.
아니나 다를까 역시 맞았다. 동정된 지 별로 오래되지 않은 균이기 때문에, 기존 HMP 데이터셋에 나타나지 않았던 것이다.
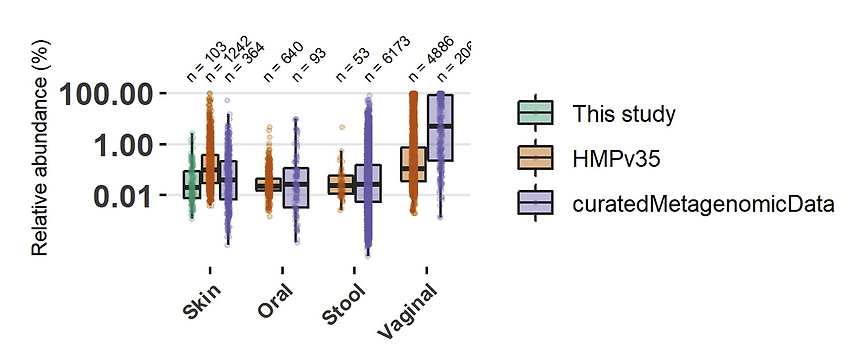
분석된 데이터셋은 16S rRNA의 V1 V3, V3V4사 사용되었으며, 현재 V3V4는 분석 중이다.
기존 HMP 16S데이터가 QIIME1기반의 OTU가 사용되었기 때문에, DADA2 결과와는 상이한 부분이 많다.
패키지는 아래 논문처럼 Opendataset의 한 종류로 사용되면 좋을 것 같다.
(Sarah Lebeer et al., Selective targeting of skin pathobionts and inflammation with topically applied lactobacilli,
Cell Reports Medicine, 2022, https://doi.org/10.1016/j.xcrm.2022.100521.)
GitHub - KitHubb/HMPData: Serve HMP data using DADA2 and SILVA database.
Serve HMP data using DADA2 and SILVA database. Contribute to KitHubb/HMPData development by creating an account on GitHub.
github.com
초기에는 V1V3의 길이를 515로 잘랐을때, 데이터 손실이 너무 커서 당혹스러웠다. 그러나 forward부분의 낮은 퀄리티를 잘라내니 평균 900 read의 샘플을 얻을 수 있었다.
기존 HMP 데이터셋을 제공하는 MicrobeDS, HM1P16SData의 경우, 평균 read 수는 약 3,300이다.
이후 길이를 450 으로 잘라냈을 때 많은 read가 퀄리티 기준을 통과했지만, ASV수가 증가된 것으로 보아 가짜-ASV의 생산이 늘어난 것으로 보인다.
자르는 길이는 이에 대해서는 정확한 기준이 없고 내 임의가 들어가있다. 그러나 450, 500의 결과가 크게 다르지 않음으로 데이터가 작은 500을 쓰는 것이 좋을 것 같다.
---
업데이트 상황
24.12.11
SILVA가 아닌 Greengenes2로 바꾸었으며, V3V4데이터를 추가하였음
