
🟦 BLAST란?
| BLAST = basic local alignment sequence tool
-BLAST는 밝혀진 지놈 또는 단백질 서열들을 데이터베이스화 해서 새로 밝혀낸 서열들의 상동성(유사성)검사를 실시하여 새로 밝혀진 서열이 어떤 기능이나 어떤 종류의 서열인지 찾아내는 것이다.
- NCBI는 command line 기반의 BLAST+프로그램을 제공한다.
| BLAST 종류 : blastn, blastp, blastx, tblastm, tblastx (NCBI)
- query(분석하고자 하는 서열)를 reference서열에 alignmet하여 어떤 친구와 가장 비슷한지 알아낸다.
- blast : query(nucleotide) ▶ reference(nucleotide)
- blastp : query(amino acid) ▶reference(amino acid)
- blastx : query(nucleotide)▶ reference(amino acid)
("▶"는 nt에서 aa로 변환 후 검색해준다는 뜻)
| Local blast란?
- local 컴퓨터(본인 컴퓨터나 서버)에 blast할 DB를 만들고 blast를 진행하는 것을 말한다.
- 온라인보다 속도가 빠르다 .
| BLAST+ 최신 버전(2022.12.19 기준)
- 다운 링크 : https://ftp.ncbi.nlm.nih.gov/blast/executables/LATEST/
🟦 설치 공식 가이드
| 공식 가이드
: https://www.ncbi.nlm.nih.gov/books/NBK52640/(영문)
BLAST+ 버전의 이름은 아래와 같다.
ncbi-blast-#.#.#+-CHIP-OS.tar.gz
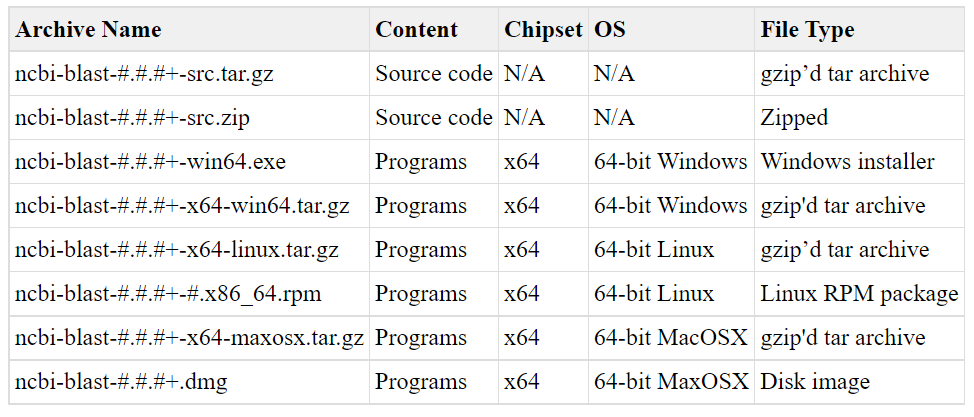
Linux기반의 64비트 컴퓨터(서버)의 경우, ncbi-blast-#.#.#+-x64-linux.tar.gz이나 ncbi-blast-#.#.#+-#.x86_64.rpm(Red Hat Package Manager) 를 다운 받아야 한다.
🟦 BLAST+ 설치
| Ubuntu / Debian
sudo apt-get install ncbi-blast+| CentOS
# download latest BLAST version
wget https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ncbi-blast-2.13.0+-x64-linux.tar.gz
# decompress
tar -zxvf ncbi-blast-2.13.0+-x64-linux.tar.gz # 이걸로 다시 해보자
# add BLAST location to system PATH
export PATH=$PATH:/home/~위치~/ncbi-blast-2.13.0+/bin
echo $PATH # Check
위치 확인
which blastn
실행 확인
blastn -help # OK
🟦 16s rRNA Database 설치
# DB directory 만들기
mkdir blast-db
cd blast-db
# 16s rRNA reference 파일 다운로드
wget https://ftp.ncbi.nlm.nih.gov/blast/db/16S_ribosomal_RNA.tar.gz
tar -zxvf 16S_ribosomal_RNA.tar.gz
# 환경변수 설정
export BLASTDB="/home/{~경로~}/blast-db/"혹은 아래 스크립트 입력
perl update_blastdb.pl --passive --decompress 16S_ribosomal_RNA
저는 perl 버전에서 오류가 계속 나는 바람에 윗 단계로 진행했다.
🟦 실제로 분석 해보기
| 분석해볼 .fa 파일 구하기
- 기존에 있는 database에서 nr_025000서열을 뽑아보자.
blastdbcmd -db {위치}/blast-db/16S_ribosomal_RNA -entry nr_025000 -out 16S_query.fa
| BLASTn 돌려보기
blastn -db {위치}/blast-db/16S_ribosomal_RNA -query 16S_query.fa -task blastn -dust no -outfmt "7 delim=, qacc sacc evalue bitscore qcovus pident sscinames" -max_target_seqs 5 > result.csv
| 결과 확인하기
# BLASTN 2.13.0+
# Query: NR_025000.1 Mycobacterium kubicae strain CDC 941078 16S ribosomal RNA, partial sequence
# Database: /home/ksy/Reference/blastdb/16S_ribosomal_RNA
# Fields: query acc., subject acc., evalue, bit score, % query coverage per uniq subject, % identity, subject sci names
# 5 hits found
NR_025000.1,NR_025000,0.0,2383,100,100.000,Mycobacterium kubicae
NR_025000.1,NR_028940,0.0,2334,100,99.243,Mycobacterium palustre
NR_025000.1,NR_125568,0.0,2320,100,98.940,Mycobacterium europaeum
NR_025000.1,NR_025760,0.0,2302,100,98.637,Mycobacterium parascrofulaceum
NR_025000.1,NR_118110,0.0,2302,100,98.637,Mycobacterium parascrofulaceum ATCC BAA-614
🚨만약 Warning: [blastn] Taxonomy name lookup from taxid requires installation of taxdb database with ftp://ftp.ncbi.nlm.nih.gov/blast/db/taxdb.tar.gz 가 뜬다면 taxa 파일을 다시 다운 받아야 한다.
cd {위치}/blast-db
# 다운로드
wget https://ftp.ncbi.nlm.nih.gov/blast/db/taxdb.tar.gz
tar -zxvf taxdb.tar.gz
# taxdb.btd
# taxdb.bti
# 경로
export BLASTDB=~/{위치}/blastdb/
# 다시 blastn
blastn -db {위치}/blast-db/16S_ribosomal_RNA -query 16S_query.fa -task blastn -dust no -outfmt "7 delim=, qacc sacc evalue bitscore qcovus pident" \
-max_target_seqs 5 > result.csv
🟦 Reference
- https://www.metagenomics.wiki/tools/blast/install
