- 목표: 올해 안 까지 Whole genome분석 환경 세팅하기
> QIIME2 shotgun에서 지원가능한지 알아보기
> Nextflow에서 사용가능한지 알아보기
1. MAGs으로 할 수 있는 연구
- 전반적 기능 예측
- Antibiotic resistance genes (ARGs)
- Antimicrobial peptides (AMPs)
- Pan-genome
- 유전자 변이 감지 (Metagenotyping)
- 미생물 대사 시뮬레이션
Fig. 2: Comparison of 16S rRNA sequencing and whole-metagenome sequencing (WMS) in microbiome analysis.
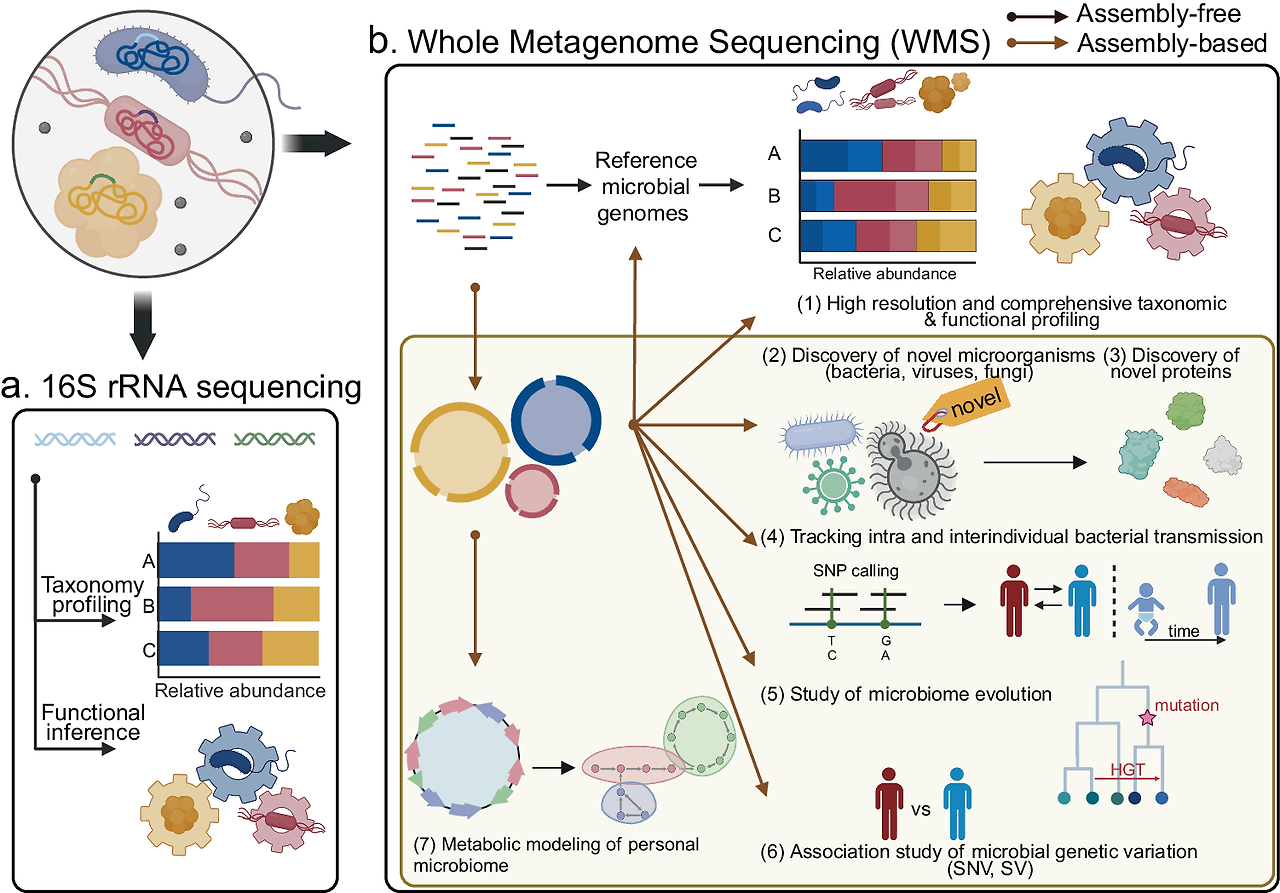
2. 사용 도구
- QC: fastqc로 확인 후 Trimmomatic, Cutadapt 사용
- Host genome removal: Bowtie2, BBmap
- Taxonomy classification: Kraken2/MetaPhlAn4 with GTDB, Pathoscope
- Assemble genome: MEGAHIT, metaSPAdes
- Assessment assembled genome: MataQUAST
- Binning: MetaBAT2, MetaWRAP, CONCOCT, Maxbin2
- Assessment Completeness and contamination: CheckM
- Predict open reading frames: Prodigal
최근 나온 대부분의 논문은 다 위의 도구 안에서 끝난다. 원하는 것을 선택하기 나름인듯하다.
3. 연구 목적에 따른 분석 도구들
1. Amplicon과 비슷하게 taxonomy만 보는 법: Kraken2, MetaPhlan4
2. 기능까지 보는 경우: Kraken2/MetaPhlan4 + HUMAnN3
3. MAGs를 만드는 경우: MEGAHIT+CONCOCT +CheckM +Prodigal
4. Pan-genome연구의 경우: MEGAHIT+ CONCOCT + CheckM + Roary
5. 미생물 변이 관찰: MEGAHIT+ CONCOCT + CheckM + StrainPhlAn
6. 대사 시뮬레이션: MEGAHIT+ CONCOCT + CheckM + RAVEN + ModelSEED
- 보통 Assenbly랑 Binning은 여러 개 도구 사용한 다음에 평가해서 가장 좋은 결괏값을 사용함.
- Taxonomy assignment도 도 Metaphlan4이랑 Kraken2 둘 다 하고, Reference DB도 여러 개 써서 Mock로 정확도(AUC) 측정해서 더 정확한 거 사용하기도 함
- 아무래도 요즘 툴이 다 좋으니, 연구에 따라 정확도 높은 도구 (혹은 그냥 인용수 제일 높은거) 쓰는 게 추세 같다.
Reference
- Kim, N., Ma, J., Kim, W. et al. Genome-resolved metagenomics: a game changer for microbiome medicine. Exp Mol Med (2024). https://doi.org/10.1038/s12276-024-01262-7
