오류 수정 : 24-01-02
- 오류 수정에 도움을 주신 분께 감사를 드립니다.
| Sankey plot이란?
각 데이터의 흐름을 나타내는 그래프로, 각 column의 다양성이 따른 범주형 데이터를 표현하기에 적합하다.

- 관련 글 : https://newsjel.ly/archives/newsjelly-report/14459
- 종합 정리 : https://www.azavea.com/blog/2017/08/09/six-sankey-diagram-tool/
- 관련 패키지 제작 : https://jhk0530.medium.com/sankey-with-shiny-505425fff443
sankey plot을 그리기에 적합한 패키지로는 ploty, ggsankey, Networkd3등이 있다. 이 중에서 Networkd3의 input파일이 제일 간단해서 이 패키지를 선택하여 시각화하였다.
| 시각화하기
library(readr)
library(tidyr)
library(dplyr)
예제 데이터
Vaccin <- data.frame(
Gender = c("F", "M", "F", "M", "F", "M", "F", "M", "F", "M", "F", "M", "F", "M", "F", "M"),
Age = c("40s","50s","20s", "40s", "20s", "50s", "30s", "30s", "50s", "40s","50s","30s", "30s", "30s", "50s", "30s"),
Vaccinated = c("Yes", "Yes","Yes", "No", "Yes","Yes", "No", "Yes", "Yes","No", "Yes", "Yes", "Yes","Yes","Yes", "No")
)
1. alluvial
alluvial 패키지를 이용해 가장 심플한 flow chart를 그려보자
dats_all <- Vaccin %>%
group_by( Gender, Age, Vaccinated) %>% # group them
summarise(Freq = n())
dats_all
# install.packages("alluvial")
library(alluvial)
alluvial( dats_all, freq=dats_all$Freq, border=NA )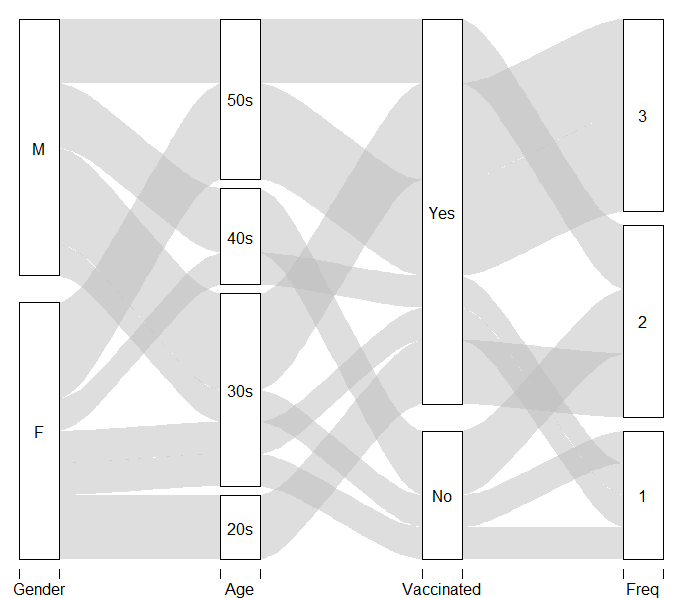
2. ggforce and ggplot2
시각화의 꽃, ggplot을 사용해 보자
library(ggforce)
dff <- Vaccin %>%
dplyr::count( Gender, Age, Vaccinated, name = "value") %>%
ggforce::gather_set_data(1:3)
dff
ggplot(dff, aes(x = x, id = id, split = y, value = value)) +
geom_parallel_sets(alpha = 0.3, axis.width = 0.1) +
geom_parallel_sets_axes(axis.width = 0.3) +
geom_parallel_sets_labels(colour = "white") +
theme_void()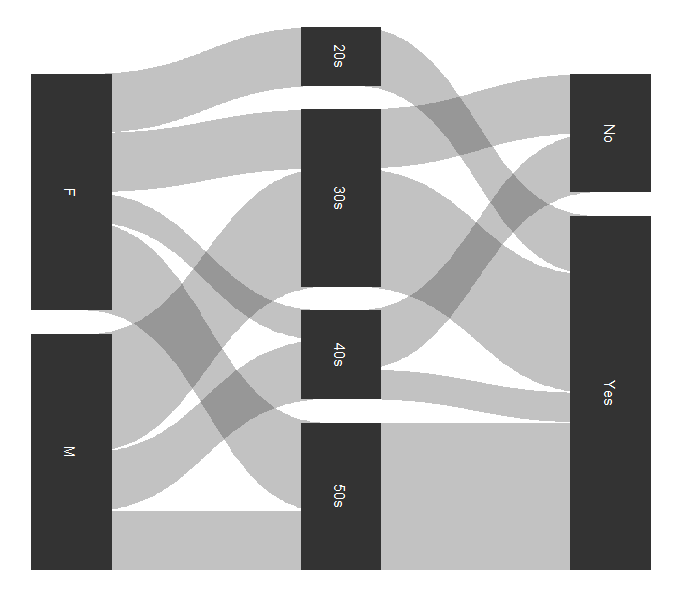
- 생각보다 그림이 깔끔하지 않다.
3. networkD3
html을 설정하여 자유로운 옵션 추가가 가능한 network3D을 이용해 보자
library(networkD3)
## 데이터 변환하기
links <-
Vaccin %>%
mutate(row = row_number()) %>%
pivot_longer(-row, names_to = "col", values_to = "source") %>%
mutate(col = col) %>%
group_by(row) %>%
mutate(target = lead(source, order_by = col)) %>%
ungroup() %>%
filter(!is.na(target)) %>%
group_by(source, target) %>%
summarise(value = n(), .groups = "drop")
links
# flow node 설정하기
nodes <- data.frame(id = unique(c(links$source, links$target)),
stringsAsFactors = FALSE)
nodes$name <- sub('_[0-9]*$', '', nodes$id)
# source와 target 노드 지정
links$source_id <- match(links$source, nodes$id) - 1
links$target_id <- match(links$target, nodes$id) - 1
# 세부 옵션
library("htmlwidgets")
# 색 지정
colourScale <-
'd3.scaleOrdinal()
.domain(["linkgrp"])
.range(["gainsboro"].concat(d3.schemeCategory20))'
# 라벨 옆 가로안에 개수 추가하기
showLabel_string <-
'function(el, x){
d3.select(el).selectAll(".node text")
.text(d => d.name + " (" + d.value + ")");}'
links
# plot 그리기
p <- sankeyNetwork(Links = links, Nodes = nodes, Source = 'source_id',
Target = 'target_id', Value = 'value', NodeID = 'name',
fontSize = 12, margin = list(t=100), colourScale = colourScale)
p <- htmlwidgets::onRender(x = p, jsCode = showLabel_string)
p
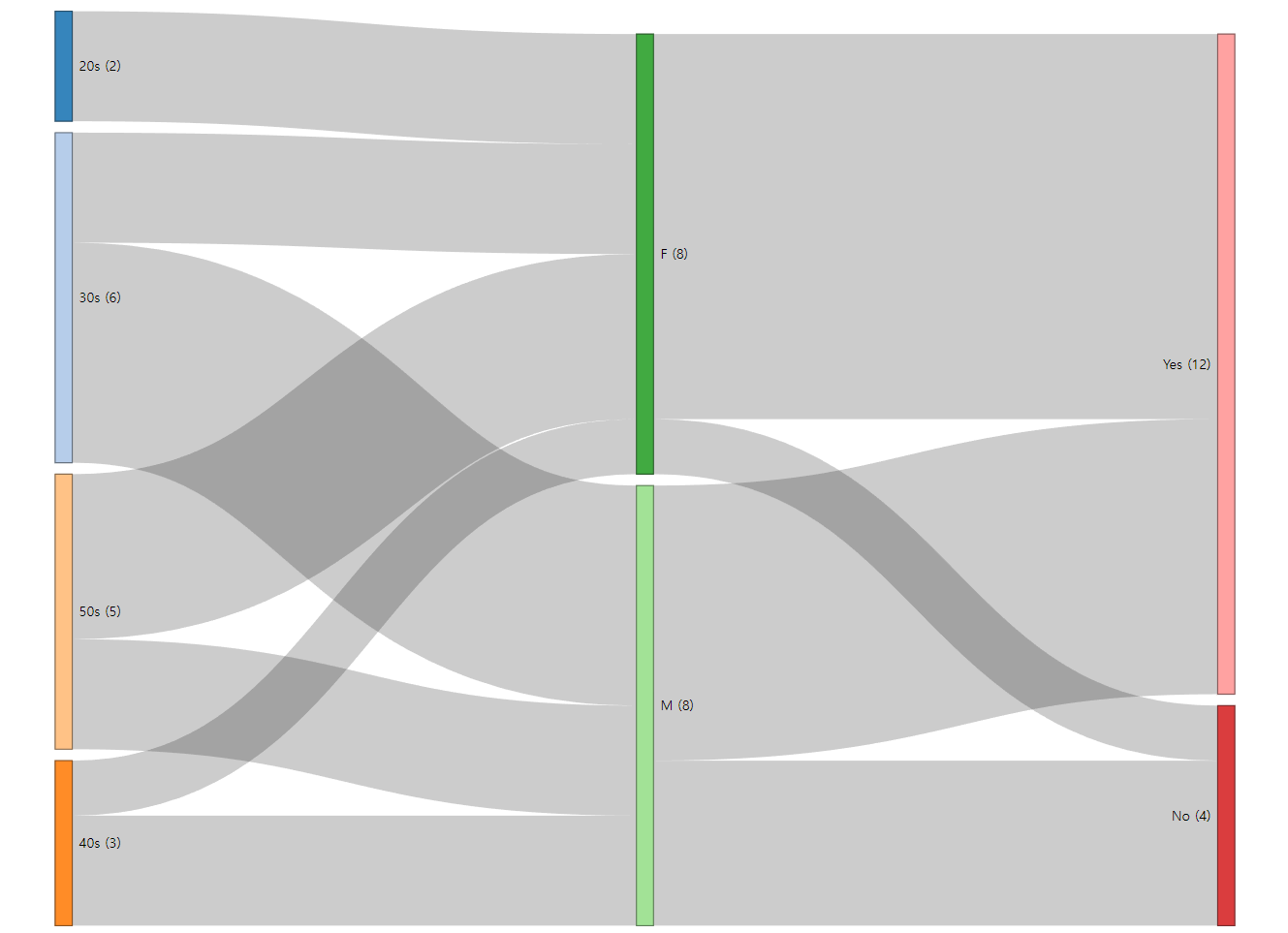
- 가장 이쁘고 여러 내용을 담을 수 있지만, 이를 추가하기 위한 옵션들이 복잡하다.
4. ggalluvial
새로 나온 ggplot 기반의 ggalluvial 패키지를 사용해 보자.
library(ggalluvial)
p <- ggplot(Vaccin,aes(axis1 = `Gender`, axis2 = `Age`, axis3 = `Vaccinated`))+
geom_alluvium(color= "black", show.legend = F) +
scale_fill_manual(values = "skyblue")+
geom_stratum()+
geom_text(stat = "stratum",
aes(label = after_stat(stratum)))+
theme_void()+
theme(legend.position = "none")
p
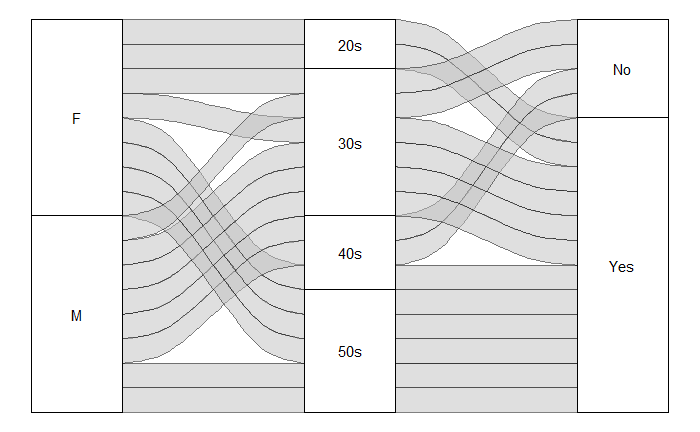
가장 깔끔한 모습을 보인다.
5. circlize
정확히는 Chord diagram 이라고 한다.
# Load the circlize library
library(circlize)
# Make the circular plot
chordDiagram(Vaccin, transparency = 0.5)
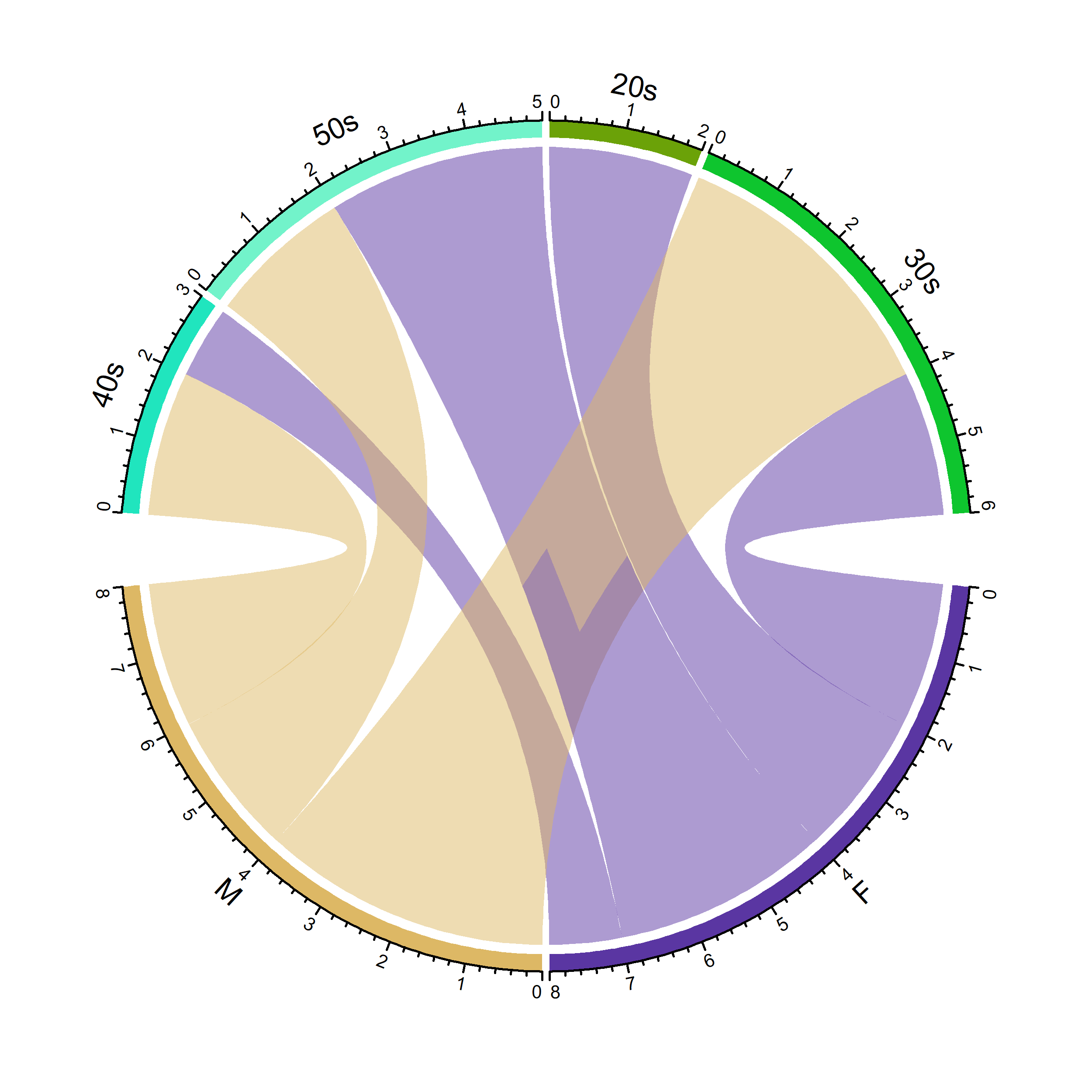
| 참고
- html문서 tistory에 첨부하기 : https://wonhwa.tistory.com/47
- https://stackoverflow.com/questions/56460820/sankey-diagram-in-r-data-preparation
sankey diagram in R - data preparation
I have the following data frame, where each patient is a row (I am showing only a sample of it): df = structure(list(firstY = c("N/A", "1", "3a", "3a", "3b", "1", "2", "1", "5", "3b"), secondY = ...
stackoverflow.com
- https://r-graph-gallery.com/sankey-diagram.html
- https://r-graph-gallery.com/123-circular-plot-circlize-package-2.html
오류 수정 : 24-01-02
- 오류 수정에 도움을 주신 분께 감사를 드립니다.
| Sankey plot이란?
각 데이터의 흐름을 나타내는 그래프로, 각 column의 다양성이 따른 범주형 데이터를 표현하기에 적합하다.

- 관련 글 : https://newsjel.ly/archives/newsjelly-report/14459
- 종합 정리 : https://www.azavea.com/blog/2017/08/09/six-sankey-diagram-tool/
- 관련 패키지 제작 : https://jhk0530.medium.com/sankey-with-shiny-505425fff443
sankey plot을 그리기에 적합한 패키지로는 ploty, ggsankey, Networkd3등이 있다. 이 중에서 Networkd3의 input파일이 제일 간단해서 이 패키지를 선택하여 시각화하였다.
| 시각화하기
library(readr)
library(tidyr)
library(dplyr)
예제 데이터
Vaccin <- data.frame(
Gender = c("F", "M", "F", "M", "F", "M", "F", "M", "F", "M", "F", "M", "F", "M", "F", "M"),
Age = c("40s","50s","20s", "40s", "20s", "50s", "30s", "30s", "50s", "40s","50s","30s", "30s", "30s", "50s", "30s"),
Vaccinated = c("Yes", "Yes","Yes", "No", "Yes","Yes", "No", "Yes", "Yes","No", "Yes", "Yes", "Yes","Yes","Yes", "No")
)
1. alluvial
alluvial 패키지를 이용해 가장 심플한 flow chart를 그려보자
dats_all <- Vaccin %>%
group_by( Gender, Age, Vaccinated) %>% # group them
summarise(Freq = n())
dats_all
# install.packages("alluvial")
library(alluvial)
alluvial( dats_all, freq=dats_all$Freq, border=NA )
2. ggforce and ggplot2
시각화의 꽃, ggplot을 사용해 보자
library(ggforce)
dff <- Vaccin %>%
dplyr::count( Gender, Age, Vaccinated, name = "value") %>%
ggforce::gather_set_data(1:3)
dff
ggplot(dff, aes(x = x, id = id, split = y, value = value)) +
geom_parallel_sets(alpha = 0.3, axis.width = 0.1) +
geom_parallel_sets_axes(axis.width = 0.3) +
geom_parallel_sets_labels(colour = "white") +
theme_void()
- 생각보다 그림이 깔끔하지 않다.
3. networkD3
html을 설정하여 자유로운 옵션 추가가 가능한 network3D을 이용해 보자
library(networkD3)
## 데이터 변환하기
links <-
Vaccin %>%
mutate(row = row_number()) %>%
pivot_longer(-row, names_to = "col", values_to = "source") %>%
mutate(col = col) %>%
group_by(row) %>%
mutate(target = lead(source, order_by = col)) %>%
ungroup() %>%
filter(!is.na(target)) %>%
group_by(source, target) %>%
summarise(value = n(), .groups = "drop")
links
# flow node 설정하기
nodes <- data.frame(id = unique(c(links$source, links$target)),
stringsAsFactors = FALSE)
nodes$name <- sub('_[0-9]*$', '', nodes$id)
# source와 target 노드 지정
links$source_id <- match(links$source, nodes$id) - 1
links$target_id <- match(links$target, nodes$id) - 1
# 세부 옵션
library("htmlwidgets")
# 색 지정
colourScale <-
'd3.scaleOrdinal()
.domain(["linkgrp"])
.range(["gainsboro"].concat(d3.schemeCategory20))'
# 라벨 옆 가로안에 개수 추가하기
showLabel_string <-
'function(el, x){
d3.select(el).selectAll(".node text")
.text(d => d.name + " (" + d.value + ")");}'
links
# plot 그리기
p <- sankeyNetwork(Links = links, Nodes = nodes, Source = 'source_id',
Target = 'target_id', Value = 'value', NodeID = 'name',
fontSize = 12, margin = list(t=100), colourScale = colourScale)
p <- htmlwidgets::onRender(x = p, jsCode = showLabel_string)
p

- 가장 이쁘고 여러 내용을 담을 수 있지만, 이를 추가하기 위한 옵션들이 복잡하다.
4. ggalluvial
새로 나온 ggplot 기반의 ggalluvial 패키지를 사용해 보자.
library(ggalluvial)
p <- ggplot(Vaccin,aes(axis1 = `Gender`, axis2 = `Age`, axis3 = `Vaccinated`))+
geom_alluvium(color= "black", show.legend = F) +
scale_fill_manual(values = "skyblue")+
geom_stratum()+
geom_text(stat = "stratum",
aes(label = after_stat(stratum)))+
theme_void()+
theme(legend.position = "none")
p

가장 깔끔한 모습을 보인다.
5. circlize
정확히는 Chord diagram 이라고 한다.
# Load the circlize library
library(circlize)
# Make the circular plot
chordDiagram(Vaccin, transparency = 0.5)

| 참고
- html문서 tistory에 첨부하기 : https://wonhwa.tistory.com/47
- https://stackoverflow.com/questions/56460820/sankey-diagram-in-r-data-preparation
sankey diagram in R - data preparation
I have the following data frame, where each patient is a row (I am showing only a sample of it): df = structure(list(firstY = c("N/A", "1", "3a", "3a", "3b", "1", "2", "1", "5", "3b"), secondY = ...
stackoverflow.com
- https://r-graph-gallery.com/sankey-diagram.html
- https://r-graph-gallery.com/123-circular-plot-circlize-package-2.html
