- 여러 통계적 테스트에서 가장 중요한것은 p-value값이 특정 값보다(대게 0.05) 높은지 낮은지 이다.
- 현재 보고자 하는 것은 피부의 각 부위별로 왼쪽과 오른쪽의 차이를 비교하고 싶다.
- 이때 permonova test를 시행한 후, 각 부위별로 p-value값을 모아서 보고자 한다.
- 사용하는 객체는 phyloseq의 예제 데이터를 사용한다 을 사용한다.
⬛ 예제 데이터
library(tidyverse)
library(phyloseq)
library(vegan)
library(glue)- Phyloseq 의 기본 예제 데이터 이용 : enterotype
- 위 데이터에서 enterotype이 각 1,2,3인 값에서 Sequencing technique(일루미나, 생어)에 따른 차이가 있는지 살펴보자
- 일단 enterotype데이터의 sample data에서 Enterotype 열의 NA값을 제거한다
data(enterotype)
enterotype
# phyloseq-class experiment-level object
# otu_table() OTU Table: [ 553 taxa and 280 samples ]
# sample_data() Sample Data: [ 280 samples by 9 sample variables ]
# tax_table() Taxonomy Table: [ 553 taxa by 1 taxonomic ranks ]
enterotype %>% sample_data() %>% data.frame() %>% .$"Enterotype" %>% unique()
# <NA> 3 2 1
enterotype <- subset_samples(enterotype, Enterotype %in% c(1,2,3) )
# phyloseq-class experiment-level object
# otu_table() OTU Table: [ 553 taxa and 271 samples ]
# sample_data() Sample Data: [ 271 samples by 9 sample variables ]
# tax_table() Taxonomy Table: [ 553 taxa by 1 taxonomic ranks ]
각 Enterotype 타입의 1,2,3을 가진 변수를 만들어 보자
types <- enterotype %>% sample_data() %>% data.frame() %>% .$"Enterotype" %>% unique()
types # 3 2 1
우리가 원하는 것은 아래와 같은 값이다.
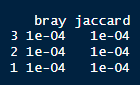
⬛ p-value를 넣을 matrix만들기
- 위 데이터는 tree데이터가 없기 때문에 "unifrac", "wunifrac" 인덱스는 사용할 수 없다.
- 일단 "bray"와 "jaccard"만을 이용하자.
# matrix
Pval <- matrix(ncol=2, nrow=length(types))
colnames(Pval) <- c("bray", "jaccard")
rownames(Pval) <- types
Pval
# bray jaccard
# 3 NA NA
# 2 NA NA
# 1 NA NA- 이렇게 비어있는 matrix를 완성했다.
⬛ 기본값 세팅
- 분석의 재현성을 위해 여러 값을 미리 정해보자
# setting
ps <- NULL
seed <- 3103
indexs <- c("bray", "jaccard")
type <- "SeqTech"
⬛ 변수 이름 추출 함수
- 아래 permenova test를 위해 한가지 함수를 만들어 보자
- 이는 변수의 이름을 사용할 수 있는 변수로 만들어 준다
# extract variable name
myfunc <- function(v1) {
deparse(substitute(v1))
}
H <- "Hello"
print(H)
"Hello"
print(myfunc(H))
# "H"이처럼 변수의 이름을 추출하는 함수이다.
⬛ 계산하기
- for문을 이용해 간단하게 반복한다.
for (i in types) {
ps <- subset_samples(enterotype, Enterotype == i )
for (index in indexs){
distan <- phyloseq::distance(ps, method = index)
set.seed(seed)
dist <- myfunc(distan)
b <- adonis2(as.formula (glue("{dist} ~ {type}")),
data=data.frame(sample_data(ps)),
permutations=9999, method=index)
Pval[i,index] <- b$`Pr(>F)`[1]
}
}
Pval
# bray jaccard
# 3 1e-04 1e-04
# 2 1e-04 1e-04
# 1 1e-04 1e-04모든 Enterotype 에서 시퀀싱 방법에 따른 차이는 모두 유의하다는 결과가 나왔다.
- 여러 통계적 테스트에서 가장 중요한것은 p-value값이 특정 값보다(대게 0.05) 높은지 낮은지 이다.
- 현재 보고자 하는 것은 피부의 각 부위별로 왼쪽과 오른쪽의 차이를 비교하고 싶다.
- 이때 permonova test를 시행한 후, 각 부위별로 p-value값을 모아서 보고자 한다.
- 사용하는 객체는 phyloseq의 예제 데이터를 사용한다 을 사용한다.
⬛ 예제 데이터
library(tidyverse)
library(phyloseq)
library(vegan)
library(glue)- Phyloseq 의 기본 예제 데이터 이용 : enterotype
- 위 데이터에서 enterotype이 각 1,2,3인 값에서 Sequencing technique(일루미나, 생어)에 따른 차이가 있는지 살펴보자
- 일단 enterotype데이터의 sample data에서 Enterotype 열의 NA값을 제거한다
data(enterotype)
enterotype
# phyloseq-class experiment-level object
# otu_table() OTU Table: [ 553 taxa and 280 samples ]
# sample_data() Sample Data: [ 280 samples by 9 sample variables ]
# tax_table() Taxonomy Table: [ 553 taxa by 1 taxonomic ranks ]
enterotype %>% sample_data() %>% data.frame() %>% .$"Enterotype" %>% unique()
# <NA> 3 2 1
enterotype <- subset_samples(enterotype, Enterotype %in% c(1,2,3) )
# phyloseq-class experiment-level object
# otu_table() OTU Table: [ 553 taxa and 271 samples ]
# sample_data() Sample Data: [ 271 samples by 9 sample variables ]
# tax_table() Taxonomy Table: [ 553 taxa by 1 taxonomic ranks ]
각 Enterotype 타입의 1,2,3을 가진 변수를 만들어 보자
types <- enterotype %>% sample_data() %>% data.frame() %>% .$"Enterotype" %>% unique()
types # 3 2 1
우리가 원하는 것은 아래와 같은 값이다.

⬛ p-value를 넣을 matrix만들기
- 위 데이터는 tree데이터가 없기 때문에 "unifrac", "wunifrac" 인덱스는 사용할 수 없다.
- 일단 "bray"와 "jaccard"만을 이용하자.
# matrix
Pval <- matrix(ncol=2, nrow=length(types))
colnames(Pval) <- c("bray", "jaccard")
rownames(Pval) <- types
Pval
# bray jaccard
# 3 NA NA
# 2 NA NA
# 1 NA NA- 이렇게 비어있는 matrix를 완성했다.
⬛ 기본값 세팅
- 분석의 재현성을 위해 여러 값을 미리 정해보자
# setting
ps <- NULL
seed <- 3103
indexs <- c("bray", "jaccard")
type <- "SeqTech"
⬛ 변수 이름 추출 함수
- 아래 permenova test를 위해 한가지 함수를 만들어 보자
- 이는 변수의 이름을 사용할 수 있는 변수로 만들어 준다
# extract variable name
myfunc <- function(v1) {
deparse(substitute(v1))
}
H <- "Hello"
print(H)
"Hello"
print(myfunc(H))
# "H"이처럼 변수의 이름을 추출하는 함수이다.
⬛ 계산하기
- for문을 이용해 간단하게 반복한다.
for (i in types) {
ps <- subset_samples(enterotype, Enterotype == i )
for (index in indexs){
distan <- phyloseq::distance(ps, method = index)
set.seed(seed)
dist <- myfunc(distan)
b <- adonis2(as.formula (glue("{dist} ~ {type}")),
data=data.frame(sample_data(ps)),
permutations=9999, method=index)
Pval[i,index] <- b$`Pr(>F)`[1]
}
}
Pval
# bray jaccard
# 3 1e-04 1e-04
# 2 1e-04 1e-04
# 1 1e-04 1e-04모든 Enterotype 에서 시퀀싱 방법에 따른 차이는 모두 유의하다는 결과가 나왔다.
