세미나 수업 2주차 논문 : Host habitat is the major determinant of the gut microbiome of fish (2021)
🟦 읽기 전
마이크로바이옴이지만, 어류의 장내 미생물에 초첨을 맞춘 연구가 새로웠다. 결론은 이미 제목에 나와있었지만, 그 결과를 얻기 위해 random forest 나 PAM같은 머신러닝 기법을 썼다고 나와있기 때문에 (내가 하고자 하는 연구의 방법적 목표임으로) 그 방법적인 부분이 궁금했다.
🟦 결론
특징적인 것만 서술하자면
- 어류의 장은 다른 척추동물의 장(인간은 소장, 대장 등)과 다르게 하나의 장만 포함하고 있다
- 다른 척추 동물 verterbrate 는 ← Firmicutes and Bacteroidetes 多 어류는 Proteobacteria 多 (포유류에서는 많으면 dysbiosis의 신호) -> 장의 unsegmented 된 구조에 의해 차이가 난다는 것을 의미
- 염분에 따른 차이 : 담수어(해수보다 양분이 부족) 는 vitamin B12 대사와 관련이 많은 Fusobacteria 多
- 식성에 따른 차이 : 해수어 Enterobacteriaceae 多 (in 육식성) / 담수어 : Clostridium/ Aeromonadaceae 多 ( 초식, 잡식)
- PAM clustering과 통계적인 분석(ANOSIM), random forest 를 통한 결과에서 어류의 Habitat가 가장 dominant한 요인으로 꼽혔다.
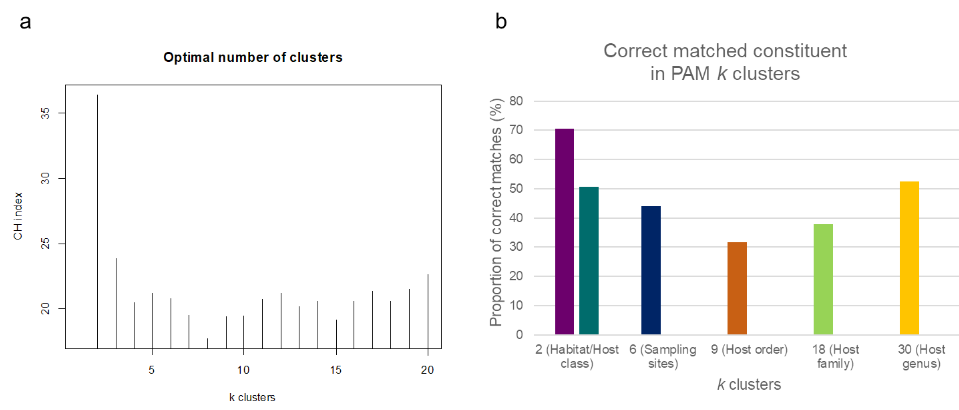
- PAM(Enterotyping | tutorial (embl.de), R 기반)은 k-means와 비슷하지만, K-means는 가상의 centroid를 기준으로 clustering하는것에 반해, PAM은 기존의 데이터 중 하나를 centroid로 잡고 clustering 한다. 일반적인 인공지능에도 잘 쓰이지 않는 분석법이라고 하는데, 왜 사용되었는지 추가적으로 알아봐야겠다
- 최적의 clustering그룹을 나눌 때에는 Calinski–Harabasz (CH) index가 사용되었다. 이는 (그룹간 분산/그룹내 분산) * (n-k/k-1) 으로 여러 k값을 대입해본 후 가장 높은 index값이 나온 cluster가 그 데이터 집단을 잘 설명한다는 뜻이다/ n; 데이터의 개수, k = cluster 수
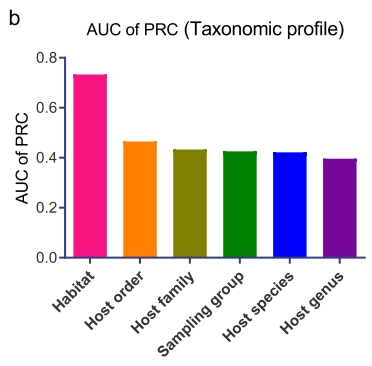
- random forest 는 Weka 3 (Java 기반)프로그램 이용했다. 여기서 AUC of PRC값은 추가적인 설명이 필요하다.
- AUC of PRC(AUPRC)
는 간단히 말해 만들어진 모델의 평가 지표이다.
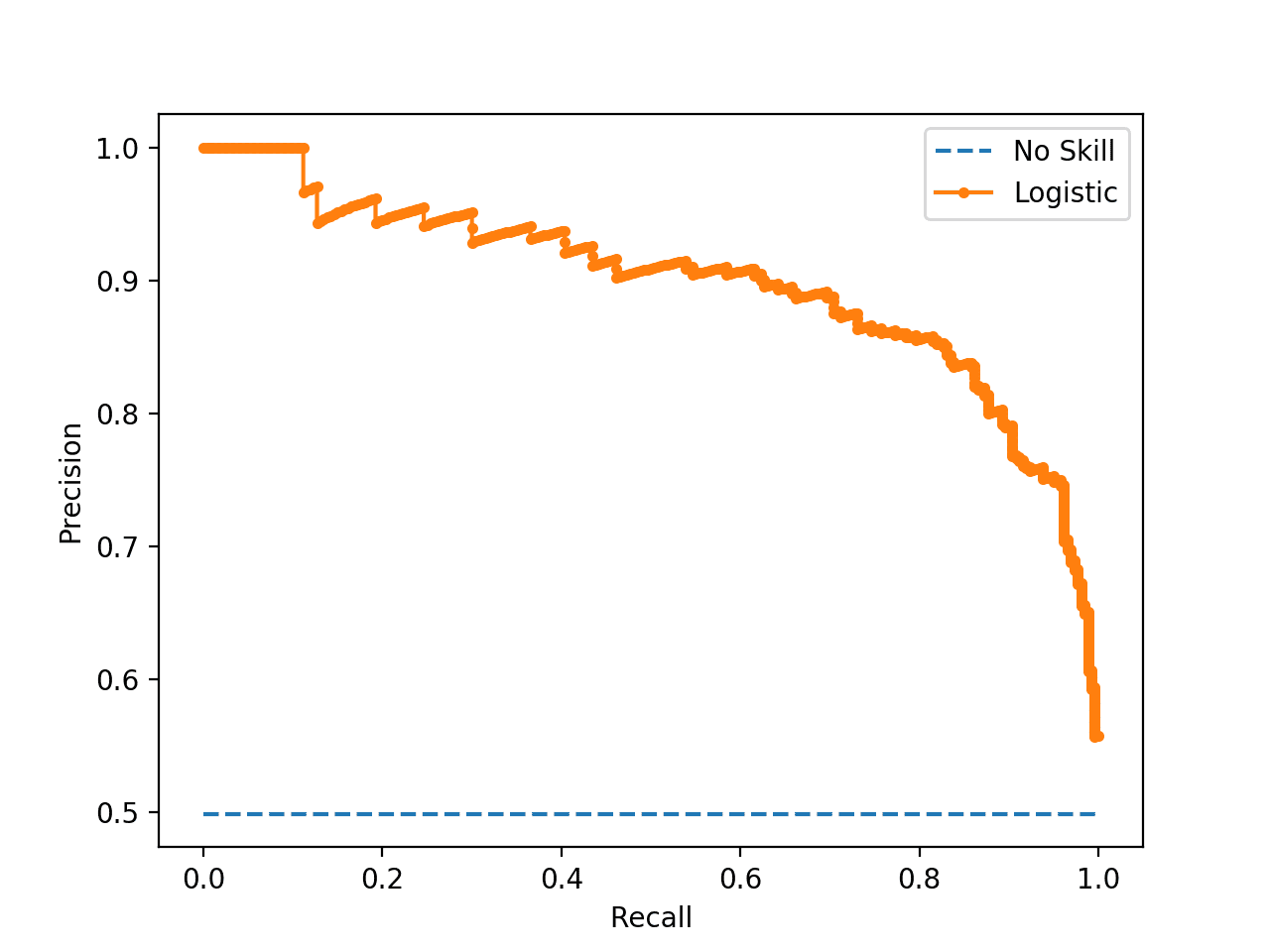
- Precision(정밀도)는 양성으로 예측된 것(양성을 양성이라고 예측 + 음성을 양성이라고 예측) 중 얼마나 많은 샘플이 진짜 양성인지
- recall(재현율)은 진짜 양성인 것(양성을 양성이라고 예측 + 양성을 음성이라고 예측)중에 얼마나 많은 샘플이 진짜 양성인지
- Precision-recall curves(PRC)은 정밀도와 재현율의 상관관계를 나타낸 곡선으로, 정밀도가 높을 수록 재현율을 낮다.
- 각 지표(임계값)을 변동시키며 그린 그림이 아래와 같은 AUC of PRC(AUPRC)를 말한다
- 이는 정밀도와 재현율이 둘다 높은 값(1)일 수록 좋기 때문에 이 그림만 본다면 recal = 0.8, precision =0.8정도가 가장 이상적으로 보인다.
🟦 기타
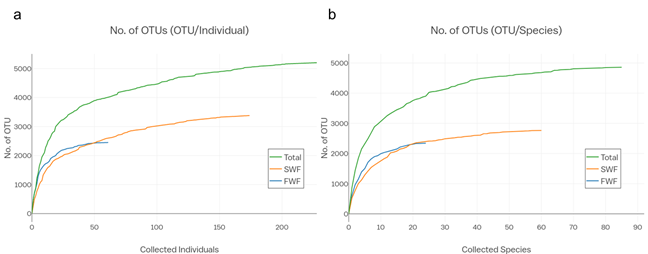
- 보통 rarefraction curve는 x 축이 sampling depth에 y축이 OTU인데, 여기는 특이하게 individuals와 species이다. => 뜻만 보다면 각 150, 60만 조사해도 존재하는 OTU는 다 나오니까 우리가 조사한 약 220여개 개체는 충분하다! 라는 의미이다. 그러나 신기해서 넣어보았다
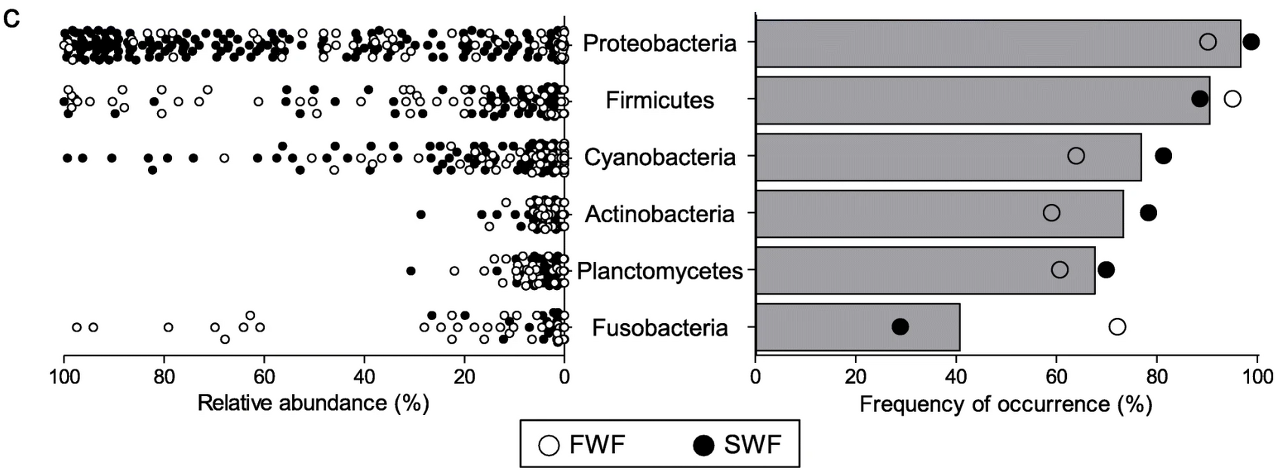
- 왼쪽은 상대적 풍부도, 오른쪽은 약 220여개의 샘플 중 몇 개의 샘플에서 각 phylum이 발견되었는지를 나타낸다. 시각적으로 잘 나타낸 자료같아서 추후에 참고하기 위해 저장해두었다. 논문에서 가장 중요한건 내용이지만 그 내용을 뒷받침 해주는 그림도 중요하다고 생각하기 때문이다.
- 발표자 분께서는 바다는 농도에 따라 담수, 해수, 기수로 나뉘는데, 위 논문에서는 기수에 사는 망뚱어같은 어류는 다루어지지 않았으며, 해수와 담수를 오가면서 성장하는 어류가 일부포함되어 있어서 아쉽다고 하셨다.
- 질문으로는 서식지(habitat)이라는 개념이 너무 모호하다고 생각하는데 결국 먹이의 차이가 아닌가? 하는 질문과 다른 포유류등도 이와 같은 결과가 나오는지, 결국 환경에 서식하는 미생물이 장내에서 발견이 적은것을 보아 장내 환경과 외부 환경이 다르다고 볼 수 있는데 왜 결론이 서식지인지 등의 질문이 있었다. 답변이 잘 기억나진 않지만 서식지라는 개념이 먹이, 염분, 농도를 총 포함하는 개념이라 논문에 habitat라고 적었을 것이라 유추한다.
🟦 느낀 점
- 읽는데 하루는 꼬박 걸린것 같다. 생각보다 모르는 단어가 너무 많고, 머신러닝 이론은 아직도 이해가 부족한 부분이 많다.
- QnA에 대한 답변을 위해선 배경지식이 중요할것 같다. 아니면 논문에 근거해 답변하는것이 좋을것 같다. 일단 공부가 제일 중요...
- 교수님께서 "논문을 읽을 때 중요한 것은 tool이 아니라 massege이다" 라고 말씀 하셨다. 나는 읽을때 어떤 tool을 가지고 했는지, 이 tool을 어떻게 사용하는지에만 집중하여 논문을 읽었던것 같아 반성하게 되었다. 또한 PICRUSt 프로그램이 사실 predicton 을 할 뿐이지 진정한 대사를 보려면 shotgun으로 분석하는 것이 맞다고 하셨다. 이것에 대해 추가적으로 알아보자..
🟦 Reference
- Kim, P.S., Shin, NR., Lee, JB. et al. Host habitat is the major determinant of the gut microbiome of fish. Microbiome 9, 166 (2021). https://doi.org/10.1186/s40168-021-01113-x
- Yosung Shim, Jiwon Chung and In-Chan Choi, "A Comparison Study of Cluster Validity Indices Using a Nonhierarchical Clustering Algorithm," International Conference on Computational Intelligence for Modelling, Control and Automation and International Conference on Intelligent Agents, Web Technologies and Internet Commerce (CIMCA-IAWTIC'06), 2005, pp. 199-204, doi: 10.1109/CIMCA.2005.1631265. / K-means 알고리즘 기반 - 클러스터링 인덱스 비교 연구
🟦 용어 정리
consortia 협력단
assemblage 집합체
deterministic 결정론적
stochastic 확률론적
predominant 우세한
autochthonous 자생적인
allochthonous 타생적인
prompt the question of ~에 대한 질문을 던지다
salinity 염분
affinity 유연, 우호, 밀접
redundancy 중복성
physiology 생리
carnivorous 육식성
herbivorous 초식성
omnivorous 잡식성
detritus 찌꺼기
interquartile 사분위수
Asterisks 별표
i.e. → 다시 말해서; 라틴어 = “that is to say” = “in other words”
unprecedented 전례 없는
elucidate 해명하다
plateau 고원
Box-and-whisker plots : box plot의 공식 명칭 (이걸 지금 알았다니..)
