이전 문제 : Translating RNA into Protein

단백질 질량 측정에 관하여
amino acid(이하 'aa')는 peptide결합으로 인해 H2O를 잃어버린다.
H2O를 잃어버리면서 결합되어있는 상태의 aa를 단백질의 residue(잔기)라고 부른다.
각각의 원자의 무게를 더하여 잔기의 무게를 제는 방법은 두 가지의 표준 방식이 있다.
1) 당일 동위원소(monoisotopic) mass
단순한 평균 질량이 아니라 동위원소(Isotope) 중에 가장 많은 비율을 차지하는 동위원소의 무게를 말함.
아래 그림처럼 무게를 측정했을때 가장 큰 피크를 그린 동위원소의 무게를 사용
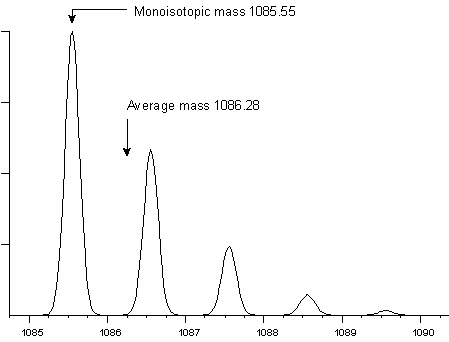
2) average mass : 동위원소들의 평균 질량을 말한다
원소의 무게를 구하기 위해 가장 많이 사용하는 것은 질량분석기(Mass spectrometry)이다.
가장 많이 사용되는 방법은 Matrix-Assisted Laser Desorption/Ionization (MALDI; 레이저를 이용해서 고체를 이온화 시키는 방법)을 통해 이온화 시키고 비행시간형 질량분석기(Time of Flight Mass Spectrometer, TOF MS)을 이용하여 질량을 계산하는 방법이다.
원소 무게의 기준은 dalton(Da)라고 불리는 atomic mass unit이다. 이는 12C를 기준으로 다른 원소의 상대적 무게를 계산한 것이다. 1DA를 가진 분자로만 이루어진 1g은 6.02×10236.02×1023 daltons으로 이루어져 있으며, 이 수를 1몰 이라고 한다.
단백질의 무게는 각 aa residue의 합 + 물 한분자의 무게이다.
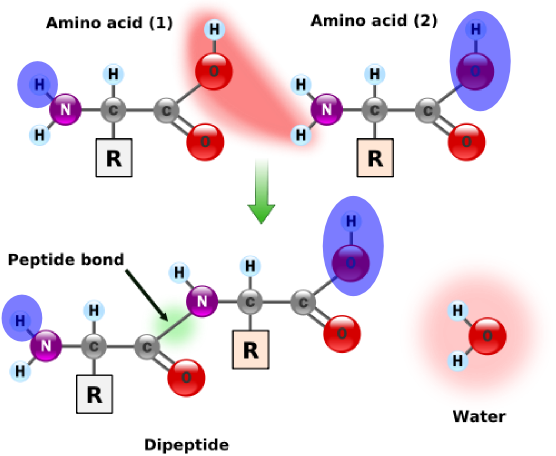
위의 이미지를 보면 peptide결합 이후에 각 aa 말단의 원자의 합이 H2O가 된다.
단순히 잔기의 합은 이 값을 포함하지 않기 때문에, 최종적인 단백질의 무게에는 잔기의 무게 + 물이 포함되어야 하는 것이다.
하지만 위 문제에서는 단순히 잔기의 무게 합만 구하고자 한다.
왜 인지는 모른다ㅋㅋ 단순히 검색해서 더 하면 된다.
자 그럼 이제 문제를 풀어보자
문제
단백질 무게를 계산하라
- input : 1000여개의 amino acid
- output : 각 aa무게의 합을 구하라
풀이
일단 각 잔기의 무게를 포함한 dictionary 파일을 만들어 주었다
molecualr_weight = {'A' : 71.03711,
'C' : 103.00919,
'D' : 115.02694,
'E' : 129.04259,
'F' : 147.06841,
'G' : 57.02146,
'H' : 137.05891,
'I' : 113.08406,
'K' : 128.09496,
'L' : 113.08406,
'M' : 131.04049,
'N' : 114.04293,
'P' : 97.05276,
'Q' : 128.05858,
'R' : 156.10111,
'S' : 87.03203,
'T' : 101.04768,
'V' : 99.06841,
'W' : 186.07931,
'Y' : 163.06333}
이제 Input 파일을 읽고 Dictionary의 keys( = aa)와 비교하여 존재하는 값의 values( = 무게)를 더한 후 반올림 해주자
with open('rosalind_prtm.txt', 'r') as f :
aa = f.read()
mass = 0
for i in aa :
mass += molecualr_weight[i]
round(mass, 3)엄청 간단하긴 하지만 다른 답변들은 진짜 상상을 초월할 만큼 더 심플했다
추천 많이 받은 답 들 by Rayan
mmt = """
A 71.03711
C 103.00919
D 115.02694
E 129.04259
F 147.06841
G 57.02146
H 137.05891
I 113.08406
K 128.09496
L 113.08406
M 131.04049
N 114.04293
P 97.05276
Q 128.05858
R 156.10111
S 87.03203
T 101.04768
V 99.06841
W 186.07931
Y 163.06333
""".split()
mmt = dict(zip(mmt[::2],map(float,mmt[1::2])))
s = "SKADYEK"
print "%.2f"%(sum(map(lambda x:mmt[x],s))+18.01528)마지막을 조금 더 쉽게 푼 풀이도 아래와 같다
print "%.2f"%(sum([ mmt[aa] for aa in s ])+18.01528)dictionary 에서 만드는 방법을 간소화할 생각은 안하고 무작정 수정한 나를 반성하며 감탄한다
대단하다
너무 오랜만에 파이썬 했더니 하나도 기억이 안난다
2년때 딕셔너리만 하는 기분이다ㅋㅋㅋ
map() 란?
# map()이란 R에서의 apply함수처럼 데이터에 지정된 함수를 적용한다
## [[in R]] ##
x <- cbind(x1 = 3, x2 = c(4:1, 2:5))
a <- apply(x, 2, sum)
a
## x1 x2
## 24 24
## [[in python]] ##
a = [1.2, 2.5, 3.7, 4.6]
a = list(map(int, a))
a
## [1, 2, 3, 4]즉 map(fload, mmt[1::2]) 란
"mnt의 두번째 값(mmn[1])부터 2개씩 뛰어서 값을 가져오는데,
이 값을 char에서 float로 변환해라" 라는 뜻
zip() 란?
# 데이터를 잘라서 짝지어주는 함수
aa = ['A', 'C', 'D', 'E', 'F', 'G']
mass = [71.03711, 103.00919, 115.02694, 129.04259, 147.06841, 57.02146]
for aa_mass in zip(aa, mass):
print(aa_mass)
## ('A', 71.03711)
## ('C', 103.00919)
## ('D', 115.02694)
## ('E', 129.04259)
## ('F', 147.06841)
## ('G', 57.02146)참고
질량분석기 : https://blog.naver.com/PostView.nhn?blogId=hyouncho2&logNo=220007347236
이전 문제 : Translating RNA into Protein

단백질 질량 측정에 관하여
amino acid(이하 'aa')는 peptide결합으로 인해 H2O를 잃어버린다.
H2O를 잃어버리면서 결합되어있는 상태의 aa를 단백질의 residue(잔기)라고 부른다.
각각의 원자의 무게를 더하여 잔기의 무게를 제는 방법은 두 가지의 표준 방식이 있다.
1) 당일 동위원소(monoisotopic) mass
단순한 평균 질량이 아니라 동위원소(Isotope) 중에 가장 많은 비율을 차지하는 동위원소의 무게를 말함.
아래 그림처럼 무게를 측정했을때 가장 큰 피크를 그린 동위원소의 무게를 사용

2) average mass : 동위원소들의 평균 질량을 말한다
원소의 무게를 구하기 위해 가장 많이 사용하는 것은 질량분석기(Mass spectrometry)이다.
가장 많이 사용되는 방법은 Matrix-Assisted Laser Desorption/Ionization (MALDI; 레이저를 이용해서 고체를 이온화 시키는 방법)을 통해 이온화 시키고 비행시간형 질량분석기(Time of Flight Mass Spectrometer, TOF MS)을 이용하여 질량을 계산하는 방법이다.
원소 무게의 기준은 dalton(Da)라고 불리는 atomic mass unit이다. 이는 12C를 기준으로 다른 원소의 상대적 무게를 계산한 것이다. 1DA를 가진 분자로만 이루어진 1g은 6.02×10236.02×1023 daltons으로 이루어져 있으며, 이 수를 1몰 이라고 한다.
단백질의 무게는 각 aa residue의 합 + 물 한분자의 무게이다.

위의 이미지를 보면 peptide결합 이후에 각 aa 말단의 원자의 합이 H2O가 된다.
단순히 잔기의 합은 이 값을 포함하지 않기 때문에, 최종적인 단백질의 무게에는 잔기의 무게 + 물이 포함되어야 하는 것이다.
하지만 위 문제에서는 단순히 잔기의 무게 합만 구하고자 한다.
왜 인지는 모른다ㅋㅋ 단순히 검색해서 더 하면 된다.
자 그럼 이제 문제를 풀어보자
문제
단백질 무게를 계산하라
- input : 1000여개의 amino acid
- output : 각 aa무게의 합을 구하라
풀이
일단 각 잔기의 무게를 포함한 dictionary 파일을 만들어 주었다
molecualr_weight = {'A' : 71.03711,
'C' : 103.00919,
'D' : 115.02694,
'E' : 129.04259,
'F' : 147.06841,
'G' : 57.02146,
'H' : 137.05891,
'I' : 113.08406,
'K' : 128.09496,
'L' : 113.08406,
'M' : 131.04049,
'N' : 114.04293,
'P' : 97.05276,
'Q' : 128.05858,
'R' : 156.10111,
'S' : 87.03203,
'T' : 101.04768,
'V' : 99.06841,
'W' : 186.07931,
'Y' : 163.06333}
이제 Input 파일을 읽고 Dictionary의 keys( = aa)와 비교하여 존재하는 값의 values( = 무게)를 더한 후 반올림 해주자
with open('rosalind_prtm.txt', 'r') as f :
aa = f.read()
mass = 0
for i in aa :
mass += molecualr_weight[i]
round(mass, 3)엄청 간단하긴 하지만 다른 답변들은 진짜 상상을 초월할 만큼 더 심플했다
추천 많이 받은 답 들 by Rayan
mmt = """
A 71.03711
C 103.00919
D 115.02694
E 129.04259
F 147.06841
G 57.02146
H 137.05891
I 113.08406
K 128.09496
L 113.08406
M 131.04049
N 114.04293
P 97.05276
Q 128.05858
R 156.10111
S 87.03203
T 101.04768
V 99.06841
W 186.07931
Y 163.06333
""".split()
mmt = dict(zip(mmt[::2],map(float,mmt[1::2])))
s = "SKADYEK"
print "%.2f"%(sum(map(lambda x:mmt[x],s))+18.01528)마지막을 조금 더 쉽게 푼 풀이도 아래와 같다
print "%.2f"%(sum([ mmt[aa] for aa in s ])+18.01528)dictionary 에서 만드는 방법을 간소화할 생각은 안하고 무작정 수정한 나를 반성하며 감탄한다
대단하다
너무 오랜만에 파이썬 했더니 하나도 기억이 안난다
2년때 딕셔너리만 하는 기분이다ㅋㅋㅋ
map() 란?
# map()이란 R에서의 apply함수처럼 데이터에 지정된 함수를 적용한다
## [[in R]] ##
x <- cbind(x1 = 3, x2 = c(4:1, 2:5))
a <- apply(x, 2, sum)
a
## x1 x2
## 24 24
## [[in python]] ##
a = [1.2, 2.5, 3.7, 4.6]
a = list(map(int, a))
a
## [1, 2, 3, 4]즉 map(fload, mmt[1::2]) 란
"mnt의 두번째 값(mmn[1])부터 2개씩 뛰어서 값을 가져오는데,
이 값을 char에서 float로 변환해라" 라는 뜻
zip() 란?
# 데이터를 잘라서 짝지어주는 함수
aa = ['A', 'C', 'D', 'E', 'F', 'G']
mass = [71.03711, 103.00919, 115.02694, 129.04259, 147.06841, 57.02146]
for aa_mass in zip(aa, mass):
print(aa_mass)
## ('A', 71.03711)
## ('C', 103.00919)
## ('D', 115.02694)
## ('E', 129.04259)
## ('F', 147.06841)
## ('G', 57.02146)참고
질량분석기 : https://blog.naver.com/PostView.nhn?blogId=hyouncho2&logNo=220007347236
