🟦 Analysis of metagenomic Data : Introdiction to PICRUSt
- 이 강의는 PICRUSt 1에 대한 설명이다
- 강의 영상 : Canadian bioinformatics Workshops : MODULE3_PICRUSt (2016)
- 강연자 : Morgan Langille
| Qualitative inference of function from taxonomy

taxa에서는 큰 차이가 보이지 않지만, 기능적으로는 많은 차이를 보인다.
| PICRUSt : a method to predict functions from 16S
| PICRUSt가 어떻게 작동하는가?
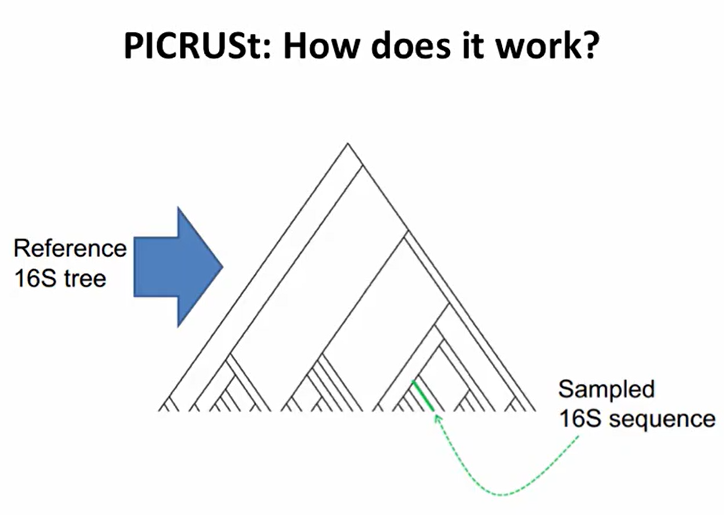
만들어진 Reference tree에 샘플의 16S를 위치시킨다.

우리 샘플이 위치한 계통수를 확대해 보자. 붉은색은 우리가 가지고 있는 유전체의 풍부도이며, 초록색은 metagenome에서 조상으로부터 예측된 기능 유전체의 풍부도이다. 이 과정을 각 기능마다 반복해서 수행산다.
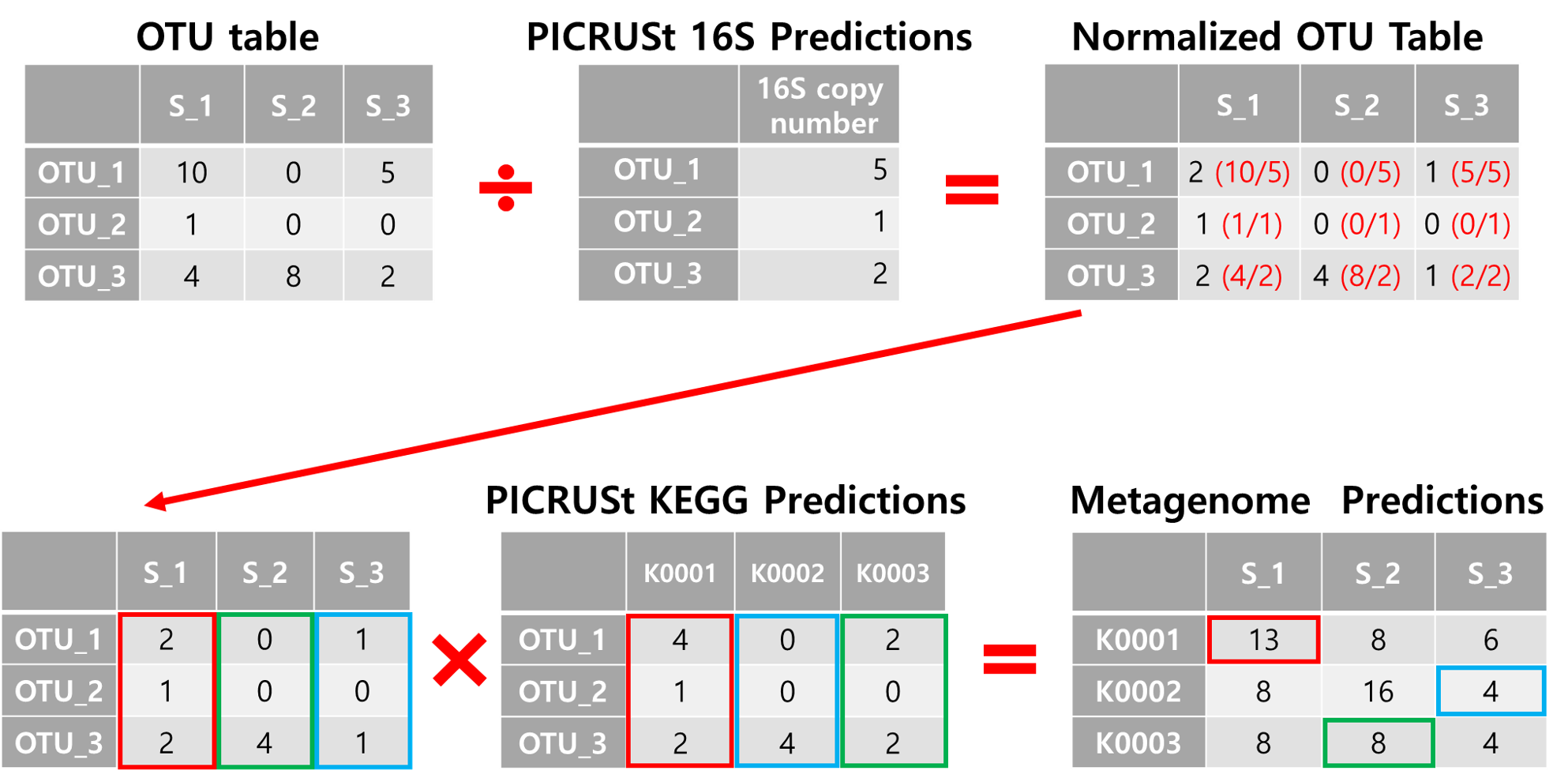
| 메타게놈 예측 👍
1. 각 OTU의 copy number를 예측하고
2. 이 copu number로 abundance를 나누고
3. 예측된 기능 유전체를 OTU table과 곱하여 최종적인 metagenome의 기능을 예측한다.
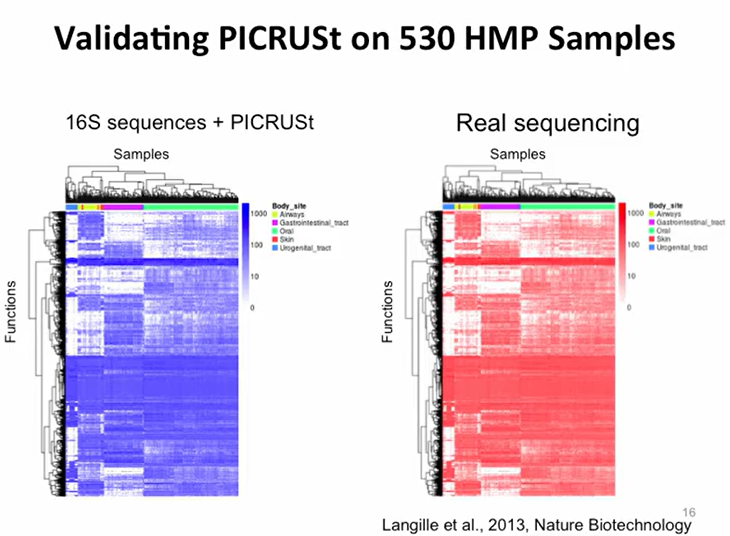
| WGS와 16S + PICRUSt의 결과 비교
| Limitations of PICRUSt (PICRUSt 1에만 해당)
- 현재 ASV가 아니라 OTU만을 를 기반으로 한다는 것 Currently based on reference OTUs only
- 현재 Greengene DB만을 reference 데이터로 참고한다는 것 Currently only works with GreenGenes DB
- 현재 18S 혹은 ITS가 아닌 16S 데이터만 분석 가능하는 점 Currently does not work with other markers(18S, ITS, etc.)
- 이상한 결과를 내놓을 수도 있음 Can include odd human function(that should be ignored)
이러한 한계점때문에, 어떤 미생물에 의해 질병이 유발된다고 말하진 않는다.

| Major steps in PICRUSr analysis
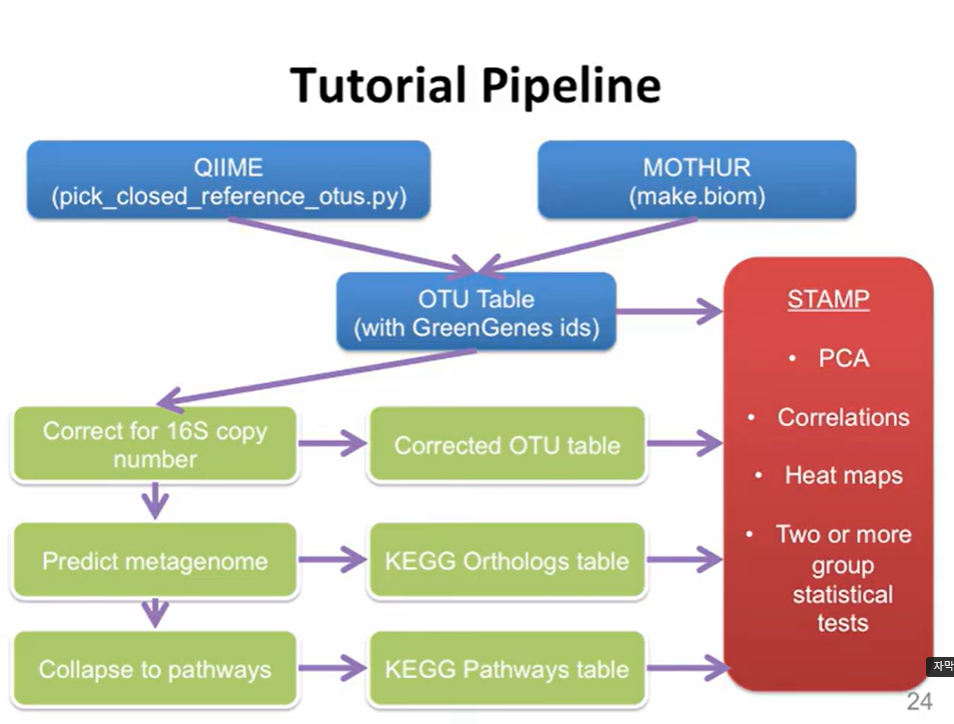
🟦 출처
- 강의 영상 : https://www.youtube.com/watch?v=0eK2ycMgVmY
- 추가적인 STAMP tutorial(화질구지) : https://www.youtube.com/watch?v=b4YI9-e1u6Q
- KEGG 데이터 시각화 with R : https://www.youtube.com/watch?v=SMBF4DyRiuo&t=167s
- 관련 심포지움 : https://www.youtube.com/watch?v=6wg-w6cCq5k
