작성 : 2022-10-24
수정 : 2023-06-04 (microbial 패키지 추가)

🟦 목표
1. Microbiome의 marker 미생물을 찾는데 많이 사용되는 LefSe 분석에 대해 알아보고
2. R을 이용하여 분석을 후 시각화해 보자
🟦 LefSe 분석이란?
LDA (linear discriminant analysis)란
이는 차원축소 방법 중 하나로, 간단히 말해 기존의 데이터의 class들을 잘 나눌 수 있는 선을 찾고 새로운 데이터가 나타났을 때 사전에 찾은 선을 기준으로 어떤 class인지 분류해 주는 알고리즘이다
(출처 : https://m.blog.naver.com/PostView.naver?isHttpsRedirect=true&blogId=ysd2876&logNo=221212453426)
LEfSe (LDA effect size) 란?
LDA를 이용하여 그룹을 비교할 때, 각 그룹에서 특징적인 미생물은 무엇이 있는지 알아내는 단계를 말한다.
예를 들어, 사람의 장과 혀의 미생물을 분석했을 때, 각 혀와 장에서만 특별히 조성이 높은 생물을 찾아내는 것이다.
분석 방법은 아래와 같은 3단계를 거친다
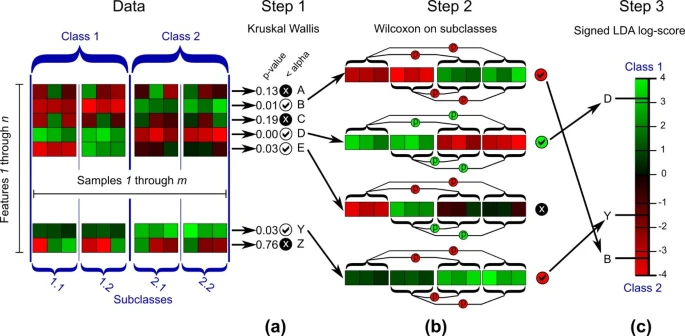
(a) Kruskal Wallis test를 통해, feature(=OTU, ASV, taxa)의 분포가 일단 각 그룹(class 1 vs class 2)에서 유의미하게 차이가 있는지 없는지 판별한다
- 여기서 B, D, E, Y가 유의하다고 판별되었다.
(b) Wlcoxon test로 class 1, 2안의 각 sub class유의 한 차이가 있는지 pairwise로(짝지어서) 비교한다
- 각각의 B, D, E에서 유의하다고 판별됨
(c) 각 feature의 effect size를 측정하기 위해 LDA가 사용된다.
- python기반으로 제작되었으며, 온라인(http://huttenhower.sph.harvard.edu/galaxy) 또는 linux환경에서 각각 분석이 가능하다.
🟦 R에서 분석해 보기 1 : microbiomeMarker
R에서 lefse분석을 하려면 lefser이나 microbiomeMarker이 필요하다.
이 중 microbiomeMarker을 사용할 것이다. 이는 2021년에 개발되었으며, 추가적인 사항은 https://yiluheihei.github.io/microbiomeMarker/index.html 여기에서 확인할 수 있다.
+) 만들어진 지 얼마 안 된 만큼 오류도 많다. 그러나 (개인적으로) bash환경에서 lefse를 분석하는 것과 비슷한 command를 쓰고, 직관적이다.
패키지 설치
if (!requireNamespace("BiocManager", quietly = TRUE)) {
install.packages("BiocManager")
}
BiocManager::install("microbiomeMarker")
library(microbiomeMarker)
library(ggplot2)
library(pheatmap)
library(tidyverse)
분석 데이터
microbiomeMarker 패키지에 있는 예제데이터를 사용할 것이다.
data(kostic_crc)
kostic_crc
#phyloseq-class experiment-level object
#otu_table() OTU Table: [ 2505 taxa and 177 samples ]
#sample_data() Sample Data: [ 177 samples by 71 sample variables ]
#tax_table() Taxonomy Table: [ 2505 taxa by 7 taxonomic ranks ]
sample_data(kostic_crc) # 데이터 구성을 확인해보자DIAGNOSIS를 보면 Healthy와 Tumor로 나뉜다.
그렇다면 우리는 건강한 그룹과 종양 그룹을 대표하는 미생물을 찾고자 할 것이다.
LefSe 분석하기
set.seed(0)
mm_test <- run_lefse( kostic_crc,
wilcoxon_cutoff = 0.01,
norm = "CPM",
group = "DIAGNOSIS",
taxa_rank = "all",
kw_cutoff = 0.01,
multigrp_strat = TRUE,
lda_cutoff = 4
)- wilcoxon_cutoff = wilcoxon 검정의 p-value
- norm = 정규화 방법, lefse 분석에서는 CPM이 default값이며, 이 방법을 추천
- group = 각 데이터에서 분석하고자 하는 데이터의 열 이름
- taxa_rank = 보고자 하는 계통수의 레벨
- kw_cutoff = kruskal wallis 검정의 p-value값
- multigrp_strat = TRUE
- lda_cutoff = lefse결과에서 특정 LDA 점수 이상만 보겠다
(출처: https://yiluheihei.github.io/microbiomeMarker/articles/microbiomeMarker-vignette.html)
- CPM : The counts per gene were normalized to CPM by dividing it by the total number of mapped reads per sample and multiplying by 1 × 10^-6
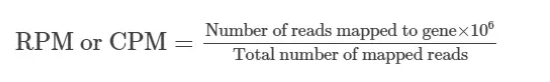
- 우리가 비교자 하고 싶은 그룹은 DIAGNOSIS이며, 각 test(wilcoxon_cutoff, kw_cutoff)의 p-value값의 한계점(alpha)을 0.01로 잡으며, LDA score가 4점 이상인 feature만 관찰하겠다는 뜻이다.
- taxa_rank에서 Species만 검색하고 싶다면 taxa_rank = "Species"로 변경하면 된다
🚨🚨Error🚨🚨
만약 아래와 같은 에러가 뜬다면 bootstrap_fraction을 기본값일 2/3보다 더 높게 설정해 보자. 4/5 -> 6/10 -> 8/10으로 늘려가는 것을 추천한다. 혹은 bootstrap_n을 너무 높게 잡았을 때, 이 값을 낮추면 해결된다.
관련 사항을 깃허브에도 문의해봤지만 본질적인 해결책은 찾지 못했다.
base::stop("infinite or missing values in 'x'")
이제 결과를 보자
marker_table(mm_test)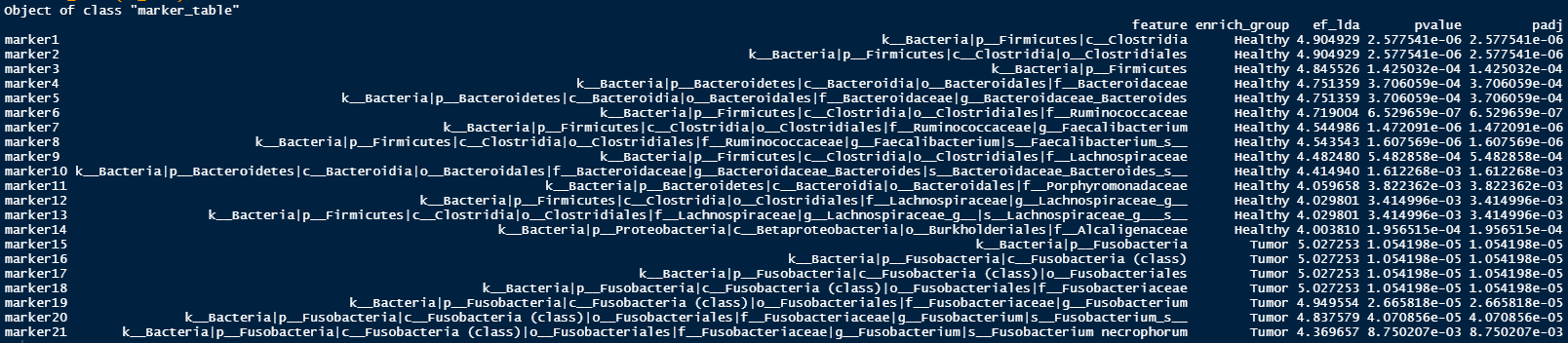
Healthy그룹을 대표하는 feature 15개와 Tumor 그룹을 대표하는 feature 6개가 검출되었다.
이를 각 LDA score를 기준으로 시각화해 본다면?
plot_ef_bar(mm_test)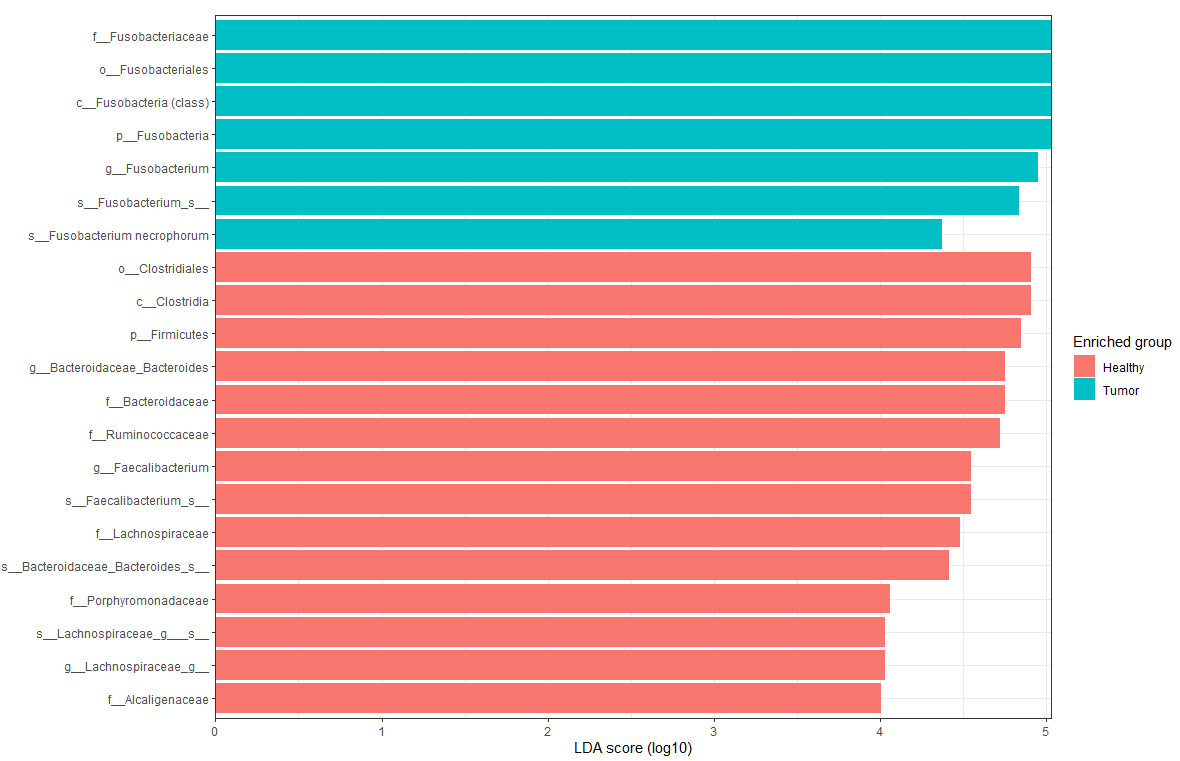
또한 cladogram으로도 볼 수 있다
plot_cladogram(mm_lefse, color = c(Healthy = "darkgreen", Tumor = "red"))
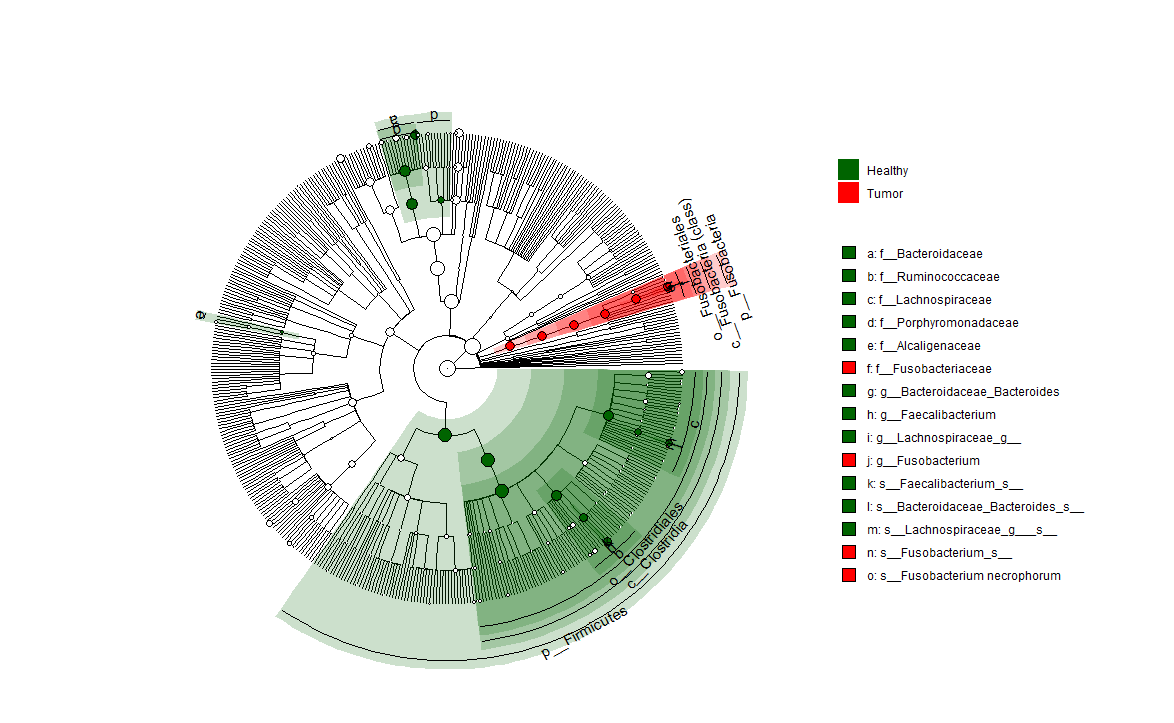
각 원의 지름은 각 taxon의 풍부도(abundance)를 나타내며, 색의 농도로 이를 표현했으며, 색은 각의 class에서 높은 풍부도를 보이는 taxon을 표시한 것이다.
🟦 LDA score구하는 다른 패키지 : microbial
# BiocManager::install("microbial")
# BiocManager::install("microbiome")
library(microbial)
library(microbiome)
# lefse 계산
res <- ldamarker(kostic_crc ,group="Group")
# plot 그리기
lefse_plot <- plotLDA(res,
group=c("Healthy","Tumor"),
lda=5,
pvalue=0.05,
color = c("red","green"),
fontsize.x = 7,
fontsize.y = 8)
lefse_plot
🟦 추가적으로 작성할 글
- 차후에는 온라인(http://huttenhower.sph.harvard.edu/galaxy/)으로 lefse분석을 돌리는 방법을 알아보자
🟦 Reference
LDA
https://m.blog.naver.com/PostView.naver?isHttpsRedirect=true&blogId=ysd2876&logNo=221212453426
LefSe
Segata, N., Izard, J., Waldron, L. et al. Metagenomic biomarker discovery and explanation. Genome Biol 12, R60 (2011). https://doi.org/10.1186/gb-2011-12-6-r60
microbiomeMarker
https://yiluheihei.github.io/microbiomeMarker/articles/microbiomeMarker-vignette.html#visualization
https://rpubs.com/mohsen/lefse_analysis_cleaned_prevalence_phyloseq
