- 작성 시작 : 2023.04.07~12
| 계통수의 구조

- 빨간 점은 root
- 초록점은 external node
- 파란 덤은 internal node
- 선은 각 node의 진화적인 변화를 선의 길이로 나타낸 것이다. 이는 아래 0.3의 scale bar로도 표시해 주었다.
| 계통수 파일의 형식
여러 형식이 있지만 가장 많이 사용되는 형식은 Newick, NEXUS, Phylip이 있다. Newick, NEXUS는 진화적 생물 데이터를 다루는 대부분의 프로그램에서 다루는 것이 가능하다. 그러나 BEAST나 MrByayes 파일은 특정 형식만을 입력 파일로만 가진다.
Newick 트리 형식
대표적인 계통수 형식인 Newick에 대해 알아보자

위와 같은 데이터를 담고 있는 계통수가 있을 때, Newick tree format은 아래와 같은 데이터로 저장된다.
((t2:0.04,t1:0.34):0.89,(t5:0.37,(t4:0.03,t3:0.67):0.9):0.59);분지 되는 그룹은 () 안에 묶고, node 간의 거리를 수로 표시해 준다. tree가 끝나면 세미콜론으로 구분을 지어준다.
| ggtree package
ggtree는 phyloseq 패키지와 호환되는 계통수 제작 패키지이다. ggtree는 R에서 사용되는 대표적인 계통수 제작패키지라고도 할 수 있으며, ggplot, tidytree, treeio, ggtreeExtra와 같이 사용된다. 아래 예시 데이터를 이용해 계통수를 그려보자.
| 계통수의 종류
ggtree를 사용할 때, 통합된 메타 패키지인 treedataverse를 다운로드하여도 되고, 아래 패키지를 일일이 다운로드하여도 된다.
treedataverse 패키지는 는 ape, dplyr, ggplot2, tidytree, treeio, ggtree, ggtreeExtra 등을 포함한다.
library(ggtreeExtra)
library(ggtree)
library(treeio)
library(tidytree)
library(ggstar)
library(ggplot2)
library(ggnewscale)
library(reshape)
ggtree에서 그릴 수 있는 트리 구조는 아래와 같다.
library(ggtree)
set.seed(2017-02-16)
tree <- rtree(50) # 50개의 노드를 가진 랜덤한 트리 생성
ggtree(tree)
ggtree(tree, layout="roundrect")
ggtree(tree, layout="slanted")
ggtree(tree, layout="ellipse")
ggtree(tree, layout="circular")
ggtree(tree, layout="fan", open.angle=120)
ggtree(tree, layout="equal_angle")
ggtree(tree, layout="daylight")
ggtree(tree, branch.length='none')
ggtree(tree, layout="ellipse", branch.length="none")
ggtree(tree, branch.length='none', layout='circular')
ggtree(tree, layout="daylight", branch.length = 'none')
또한 세부적인 옵션 추가로 아래와 같은 그림으로도 그릴 수 있다.
ggtree(tree) + scale_x_reverse()
ggtree(tree) + coord_flip()
ggtree(tree) + layout_dendrogram()
ggplotify::as.ggplot(ggtree(tree), angle=-30, scale=.9)
ggtree(tree, layout='slanted') + coord_flip()
ggtree(tree, layout='slanted', branch.length='none') + layout_dendrogram()
ggtree(tree, layout='circular') + xlim(-10, NA)
ggtree(tree) + layout_inward_circular()
ggtree(tree) + layout_inward_circular(xlim=15)
| phyloseq 데이터를 이용해 계통수 그려보기
아래의 phyloseq형태의 데이터는 QIIME2 tutorial에 사용되는 moving picture데이터를 phyloseq으로 변환한 것이다. 이 데이터는 사람의 각 손과 장, 양 손바닥의 마이크로바이옴을 비교하였다.
데이터 읽어오기
library(ggtreeExtra)
library(ggtree)
library(treeio)
library(tidytree)
library(ggstar)
library(ggplot2)
library(ggnewscale)
library(TDbook)
library(reshape)
## 파일 불러오기 ##
ps <- readRDS("./ps.rds")
ps
# phyloseq-class experiment-level object
# otu_table() OTU Table: [ 770 taxa and 34 samples ]
# sample_data() Sample Data: [ 34 samples by 8 sample variables ]
# tax_table() Taxonomy Table: [ 770 taxa by 7 taxonomic ranks ]
# phy_tree() Phylogenetic Tree: [ 770 tips and 768 internal nodes ]
## read count to relative abundance ##
ps.rel <- transform_sample_counts(ps, function(x) x/sum(x) ) # 770 taxa
## 데이터 일부만 추출 ##
ps.rel.bac <- subset_taxa(ps.rel, Kingdom == "Bacteria" &
Phylum != "Unclassified Bacteria") # 748 taxa
myTaxa = names(sort(taxa_sums(ps.rel.bac), decreasing = TRUE)[1:50])
ps.50 = prune_taxa(myTaxa, ps.rel.bac)
# phyloseq-class experiment-level object
# otu_table() OTU Table: [ 50 taxa and 34 samples ]
# sample_data() Sample Data: [ 34 samples by 8 sample variables ]
# tax_table() Taxonomy Table: [ 50 taxa by 7 taxonomic ranks ]
# phy_tree() Phylogenetic Tree: [ 50 tips and 49 internal nodes ]데이터의 크기를 줄이기 위해 Top 50 taxa만 관찰해 보자
계통수 그리기
기본적인 계통수를 그려보자. extrenal node의 색은 Phylum별로 구분하였다. 이때 기본적인 OTU단위로 node가 생성된다.
ggtree(ps.50, layout="circular", open.angle=10, ) +
geom_tippoint(mapping=aes(color=Phylum), size=0.5, show.legend=T)
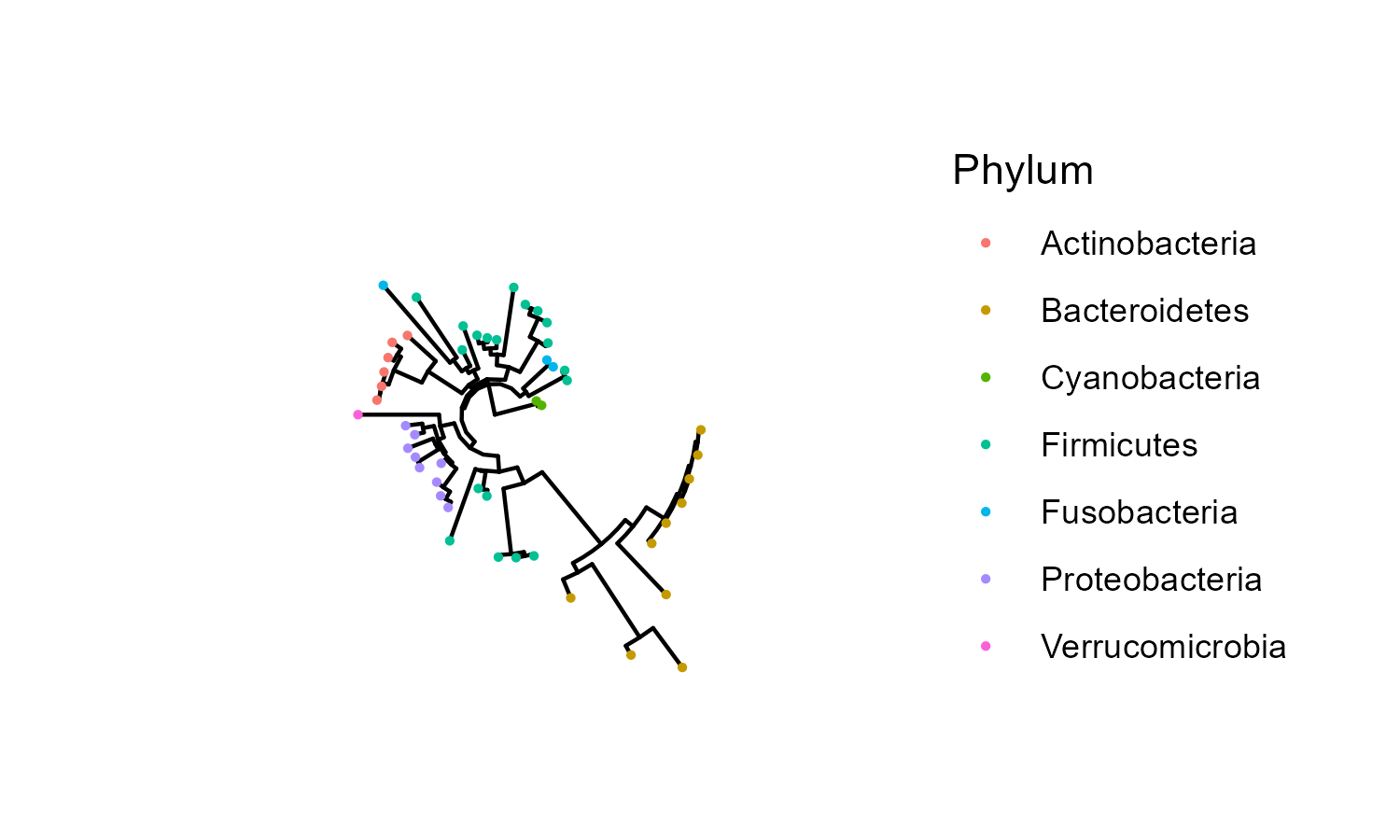
계통수 내에서 Phylum마다 거리가 떨어져 있는 모습을 볼 수 있다. 추가적으로 각 OTU의 relative abundance의 정보를 boxplot으로 표현해 보자.
p <- ggtree(ps.50, layout="circular", open.angle=10, ) +
geom_tippoint(mapping=aes(color=Phylum), size=0.5, show.legend=F) +
geom_fruit(geom=geom_boxplot,
mapping = aes(
y=OTU,
x=Abundance,
fill=Phylum),
size=.2,
outlier.size=0.5,
outlier.stroke=0.08,
outlier.shape=21,
axis.params=list(
axis = "x",
text.size = 1.8,
hjust = 1,
vjust = 0.5,
nbreak = 3),
grid.params=list())
p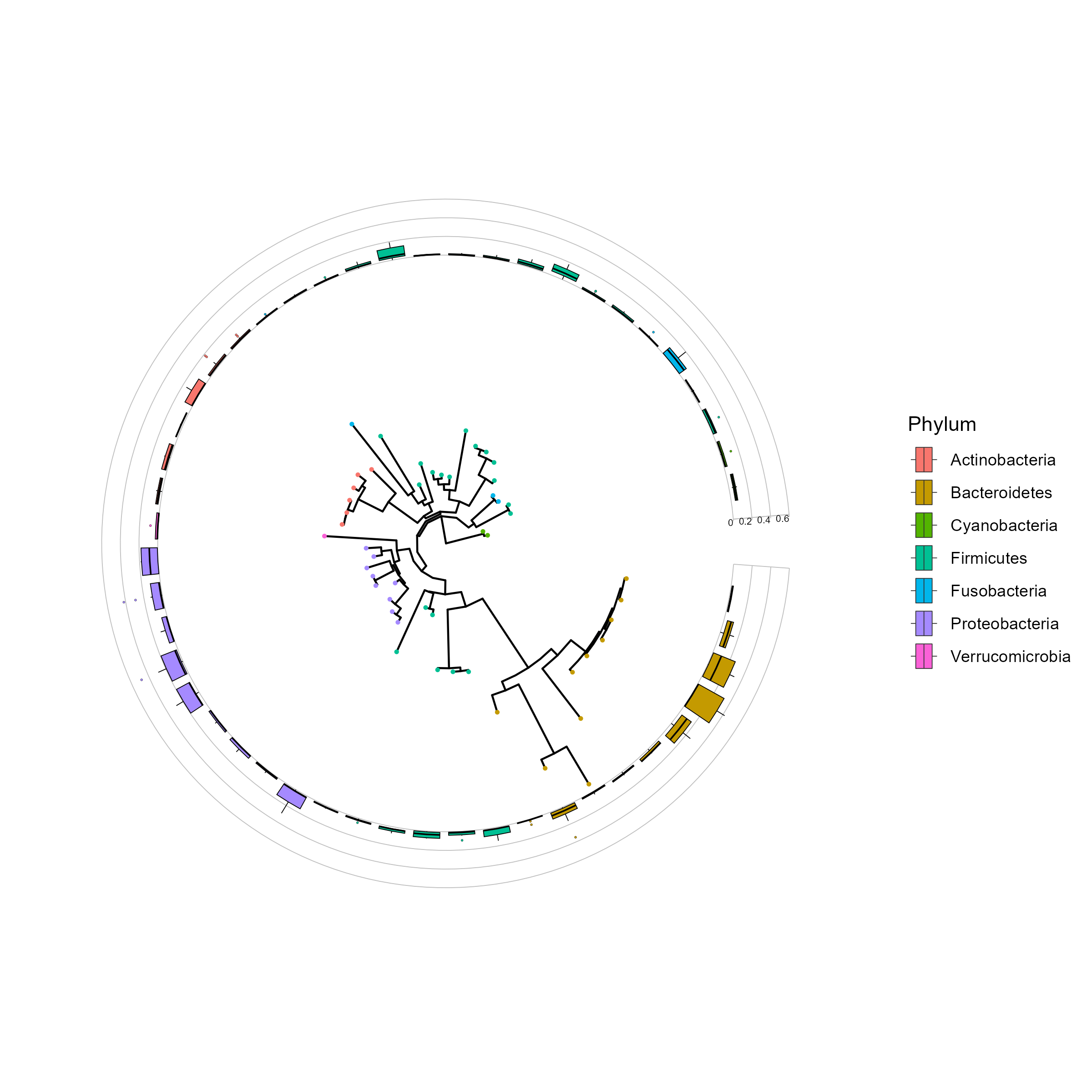
이 데이터의 샘플링 구역은 gut, tongue, right palm, left palm이다. otu가 각 샘플링 부위에서 얼마나 풍부하게 존재하는지 알아보기 위해 타일 형태의 그림(geom_tile)을 추가하여 살펴보자.
이때 ggnewscale::new_scale_fill()을 적은 후 그 밑에 그리고 싶은 내용을 추가한다.
p2 <- p + new_scale_fill() +
geom_fruit(geom=geom_tile,
mapping=aes(y=OTU,
x = `body.site`,
fill=`body.site`,
alpha = Abundance),
color = "grey30", offset = 0.04, size = 0.02) +
scale_alpha_continuous(range=c(0, 1),guide=guide_legend(keywidth = 0.3, keyheight = 0.3, order=5))
p2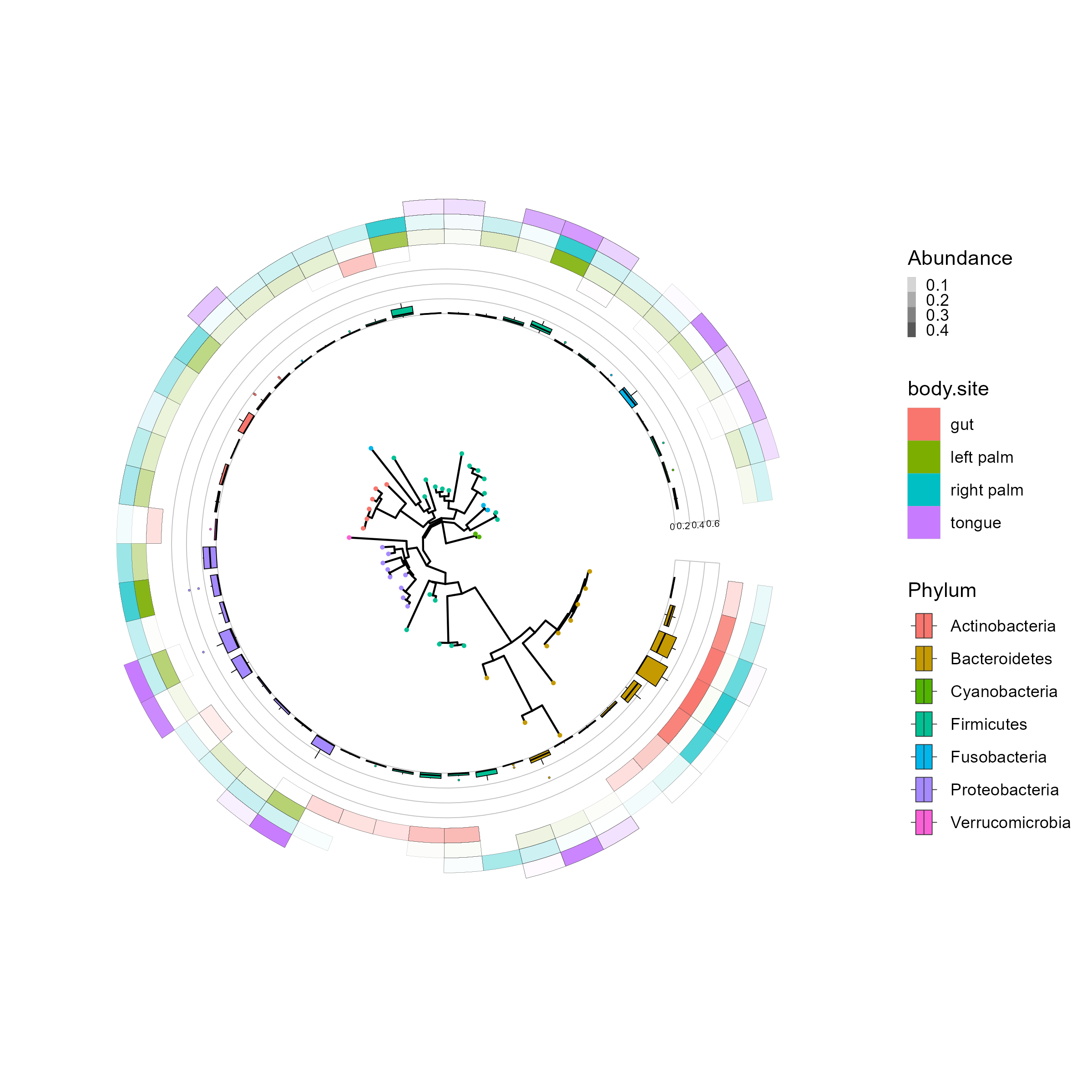
그림을 보면 Bacteroidetes Phylum에 속하는 OTU는 장과 오른손바닥에 풍부하게 존재하는 것을 알 수 있다. 이렇게 하나의 그림에 종합적인 정보를 표현할 수 있다.
각 node에 OTU ID나 Phylum정보를 붙이고 싶다면 geom_tiplab(aes(label= {otu혹은 taxa level})) 을 사용한다.
p2 + geom_tiplab(aes(label=Genus), size = 3)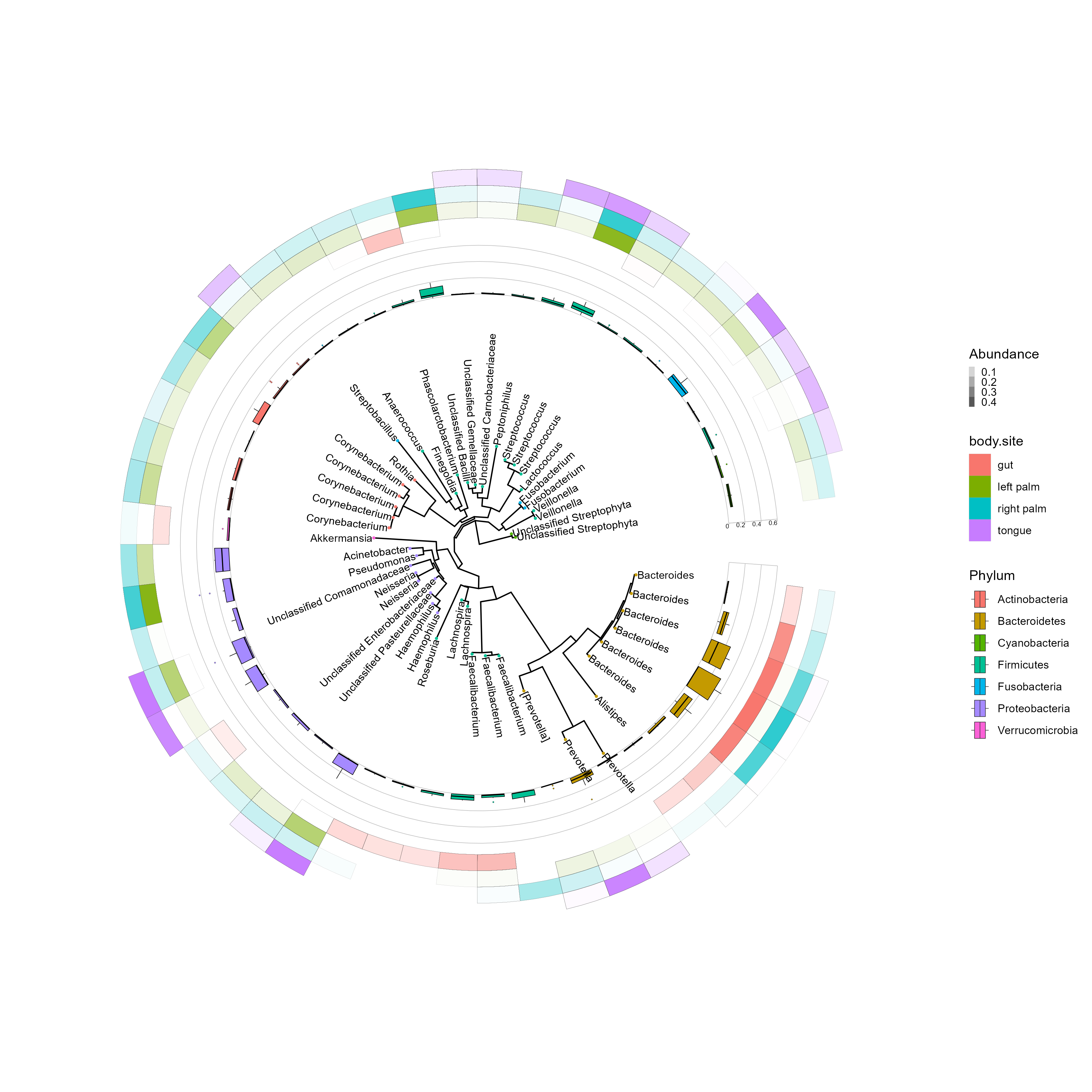
아래처럼 트리의 구조를 바꾸어도 동일한 내용을 출력할 수 있다.
ggtree(ps.50, layout="rec", open.angle=10, ) +
geom_tippoint(mapping=aes(color=Phylum), size=0.5, show.legend=F) +
geom_fruit(geom=geom_boxplot,
mapping = aes(
y=OTU,
x=Abundance,
fill=Phylum),
size=.2,
outlier.size=0.5,
outlier.stroke=0.08,
outlier.shape=21,
axis.params=list(
axis = "x",
text.size = 1.8,
hjust = 1,
vjust = 0.5,
nbreak = 3),
grid.params=list()) +
new_scale_fill() +
geom_fruit(geom=geom_tile,
mapping=aes(y=OTU,
x = `body.site`,
fill=`body.site`,
alpha = Abundance),
color = "grey30", offset = 0.04, size = 0.02) +
scale_alpha_continuous(range=c(0, 1),guide=guide_legend(keywidth = 0.3, keyheight = 0.3, order=5))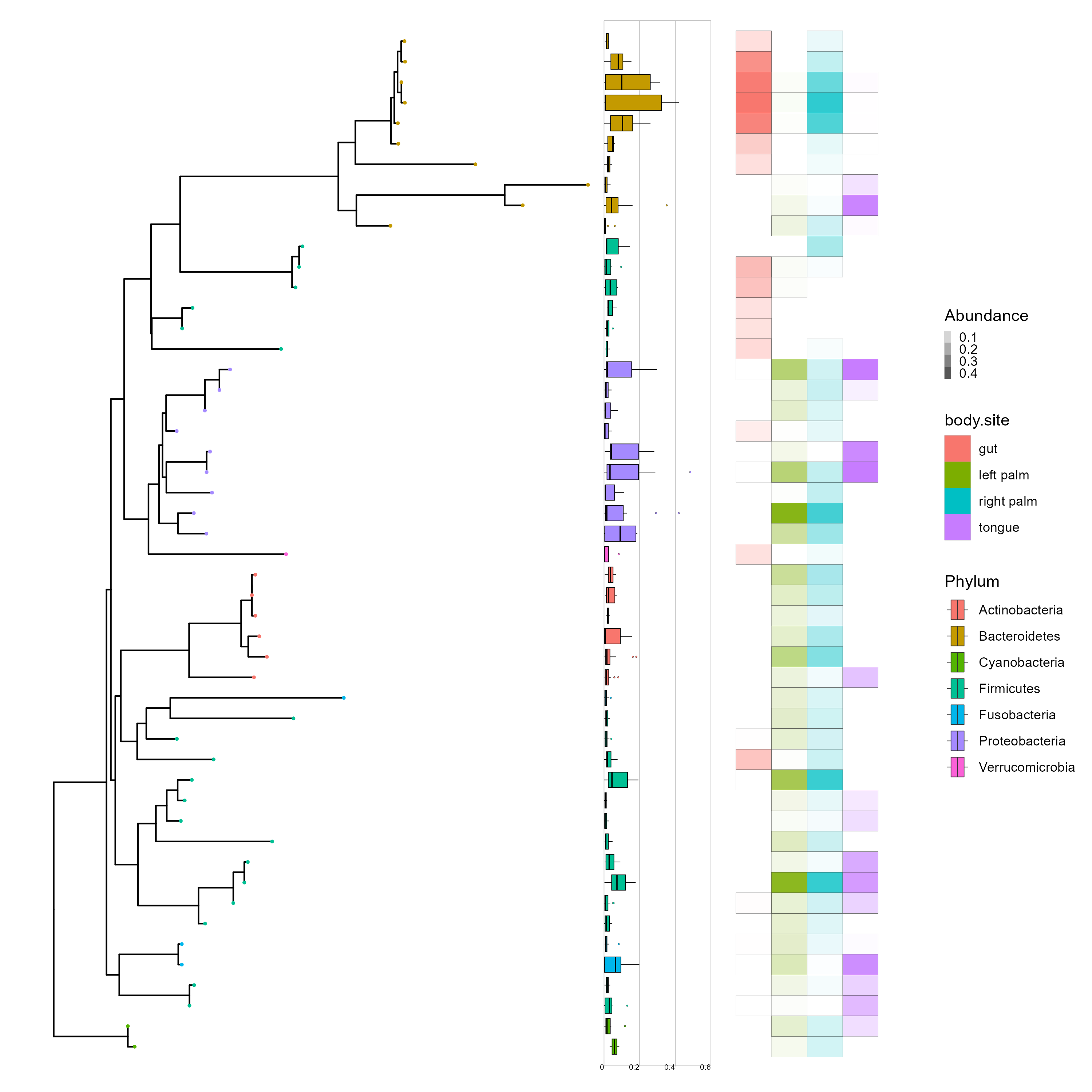
| Phyloseq 데이터가 아닌 트리 데이터에 메타데이터 추가하기
ggtree는 꼭 phyloseq데이터를 input으로 받지 않는다. 여러 형식의 tree파일을 ggtree개체로 만든 후에, 메타데이터를 추가고 입력하는 것도 가능하다.
메타데이터는 아래의 dt.melt 데이터 형식을 따라야 한다. 이 랜덤 한 트리의 메타데이터는 Sample_1,2,3에서 Phylum수준의 taxa 4개의 relative abundance 정보를 담고 있다.
# 예제 트리 데이터 만들기
set.seed(42)
tr <- rtree(100)
# 예제 메타 데이터 만들기
dt = data.frame(id=tr$tip.label,
Sample_1=abs(rnorm(100)),
Sample_2=abs(rnorm(100)),
Sample_3=abs(rnorm(100)),
Phylum=c(rep("Phylum_1",33),rep("Phylum_2",20),
rep("Phylum_3",10), rep("Phylum_4",37)))
dt.melt <- melt(dt)
dt.melt
# id Phylum variable value
# 1 t5 Phylum_1 Sample_1 1.3664669
# 2 t33 Phylum_1 Sample_1 0.3942079
# 3 t49 Phylum_1 Sample_1 0.3163953
# 4 t73 Phylum_1 Sample_1 0.8490450
# 5 t29 Phylum_1 Sample_1 1.0064365
# 6 t76 Phylum_1 Sample_1 2.2754909
100개의 노드로 이루어진 랜덤 트리를 살펴보자.
ggtree(tr, layout="circular")
위 계통수를 phyloseq예제 데이터와 비슷하게 시각화해보자.
그러나 tr 파일은 phyloseq데이터처럼 sample_data를 포함하고 있지 않은 샘플이다. 이런 파일은 이전에 만들어둔 dt.melt파일을 geom_fruit() 함수 안에 넣어서 계통수의 정보를 추가해 준다.
ggtree(tr, layout="circular", open.angle=10) +
geom_fruit(data=dt.melt, # 메타데이터 추가
geom=geom_boxplot, # boxplot 시각화
mapping = aes(
y=id,
x=value,
fill=Phylum), # Phylum에 따라 box 색 지정
size=.2,
outlier.size=0.5,
outlier.stroke=0.08,
outlier.shape=21,
axis.params=list(
axis = "x",
text.size = 1.8,
hjust = 1,
vjust = 0.5,
nbreak = 3),
grid.params=list()) +
new_scale_fill() +
geom_fruit(data=dt.melt,
geom=geom_tile, # 타일 형식으로 시각화
mapping=aes(y=id,
x = variable,
fill=variable, # 채울 색
alpha = value), # 투명도 지정
color = "grey50", offset = 0.04,size = 0.02)+ # tile 경계선
scale_alpha_continuous(range=c(0, 1),
guide=guide_legend(keywidth = 0.3,
keyheight = 0.3, order=5))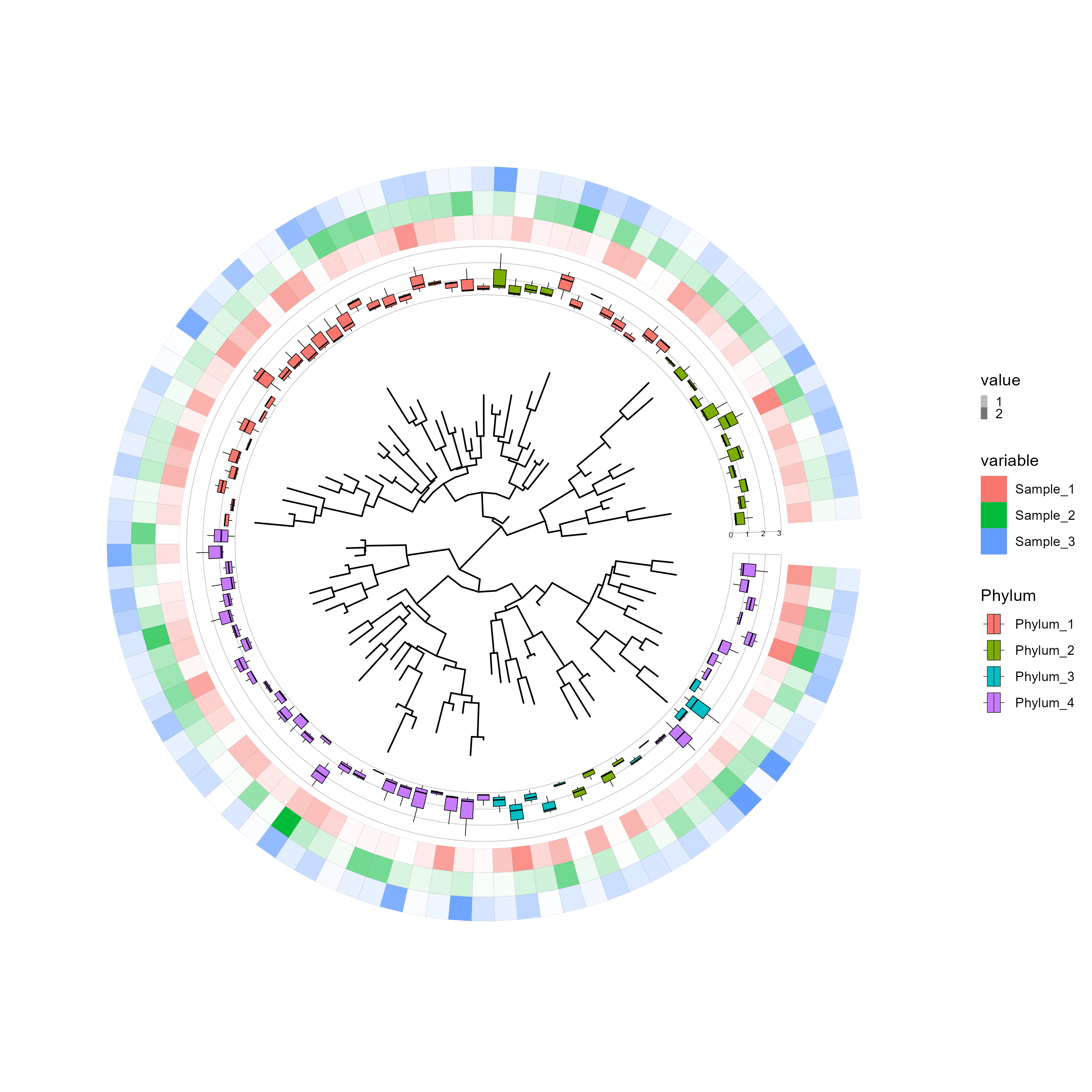
| 출처
- https://yulab-smu.top/treedata-book/
