| 계통수 시각화 프로그램 중 가장 많이 사용되는 Top 5
계통수 파일이 형식은 대부분의 소프트웨어에서 사용 가능하다. 정말 많은 도구가 있지만 그중에서 가장 널리 쓰이는 5개의 툴을 가지고 왔다.
아래 예제데이터인 phyloseq의 계통수를 가지고 각 프로그램으로 계통수를 그려보자. 예제 데이터는 qiime2의 moving-picture 데이터를 phyloseq개체로 변환한 것이다.
library(ape)
library(phyloseq)
ps <- readRDS("./ps.rds")
tree <- phy_tree(phyloseq)
ape::write.tree(tree, "./ps_tree.tree") # Nexus
ape::write.tree(tree, "./ps_tree2.tre") # Newick
1. MEGA [1]
MEGA는 계통수의 시각화뿐만 아니라 제작하는 것도 가능하다. fasta파일만 넣으면 alignment부터 여러 알고리즘으로 제작된 계통수를 만들 수 있다. 또한 만들어진 계통수를 tree explorer창을 통해 쉽게 편집이 가능하다. 트리의 input 형식으로는 newick, phylip, mega, nexus와 MEGA에 사용되는 mtsx, mtx가 있다. 만약 다른 형식을 mtsx로 변환하고 싶다면 링크에 접속하여 업로드 후 바꿀 수 있다. 다양한 OS를 지원하고 GUI환경으로 마우스로 편하게 조작이 가능해서 초보자의 경우 이 프로그램을 추천한다. 👍
- MEGA11로 계통수 제작하는 방법 : 블로그 링크
- 관련 강의 영상(영) : 서열 다운로드부터 계통수 그리기까지, 2
MEGA 에서 계통수 보기
1. MEGA11 다운로드 : MEGA 홈페이지
2. File - Open A File - ps_tree2.tre 클릭
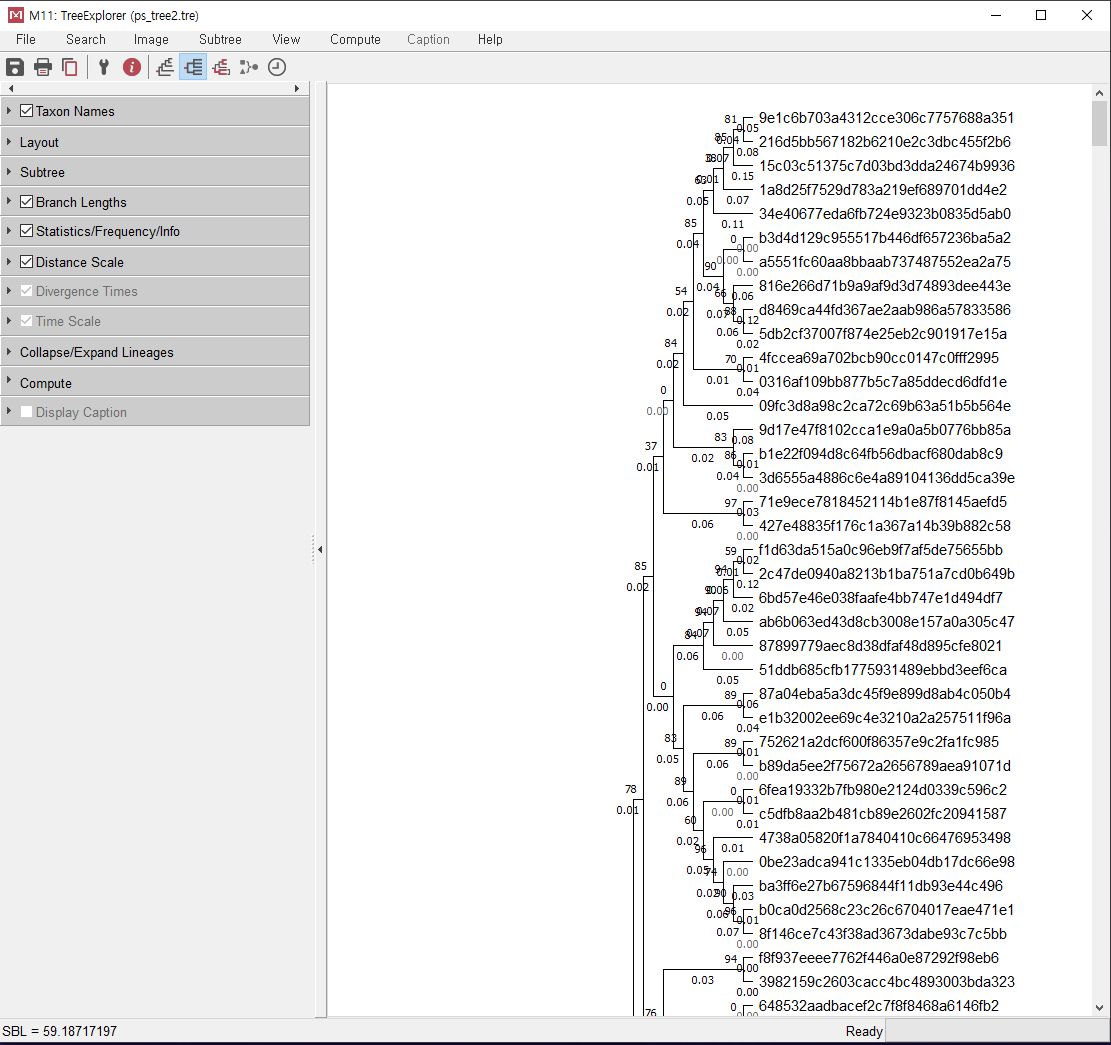
Phyloseq데이터에 저장된 OTU ID로 라벨링 된 것을 볼 수 있다.
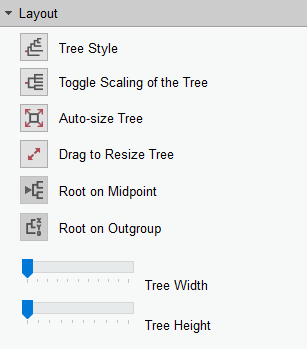
왼쪽 상단의 Layout옵션을 통해서 시각적인 옵션을 바꿀 수 있다.
2. Dendroscope [2]
자바로 작성된 계통수 작성프로그램으로, 큰 크기의 계통수를 시각화하기 좋은 프로그램이다. 위 프로그램은 윈도우, 맥, 리눅스를 지원한다. 트리는 newick나 nexus 형식으로 내보낼 수 있다.
- 다운로드 링크 : https://software-ab.cs.uni-tuebingen.de/download/dendroscope3/welcome.html(현재 ver 3)
- 매뉴얼 : https://software-ab.cs.uni-tuebingen.de/download/dendroscope/manual.pdf
Dendroscope 에서 계통수 보기
1. 다운로드 링크에서 윈도우 전용 Dendroscope_windows-x64_3_8_8파일 다운로드
2. 프로그램 설치
3. File - Open - ps_tree2.tre 클릭
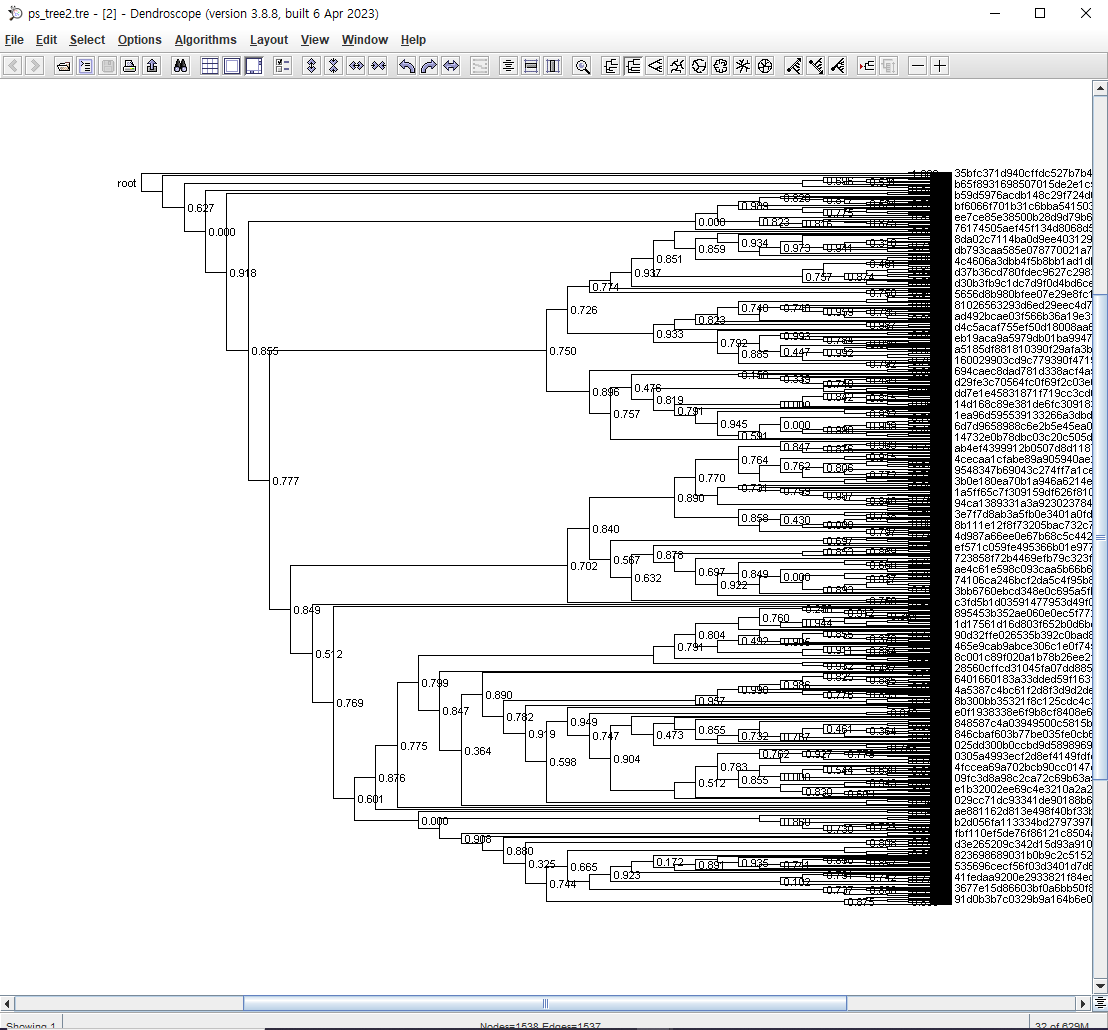
UI가 너무 촌스러운게 단점이지만 프로그램이 가벼워서 큰 데이터를 시각화하기에 알맞을 것 같다.
3. FigTree [3]
계통수의 시각화 프로그램으로 다양한 형식(PHYLIP, IQTree, RAxML, PAML, BEAST)을 input파일로 받는다.
FigTree도 MEGA처럼 다양한 OS를 지원하고 GUI환경으로 마우스로 편하게 조작이 가능하다.
- 관련 정보 위키 : 인코덤
- 홈페이지 : http://tree.bio.ed.ac.uk/software/figtree/
FigTree에서 계통수 보기
1. 자바 다운로드(필수) : https://www.java.com/ko/download/
2. FigTree 다운로드(최신버전 1.4.5) : https://github.com/rambaut/figtree/releases
3. FigTree.v1.4.3.zip 압축 풀기
4. 압축 풀린 폴더 안에 있는. exe 파일을 실행한다.
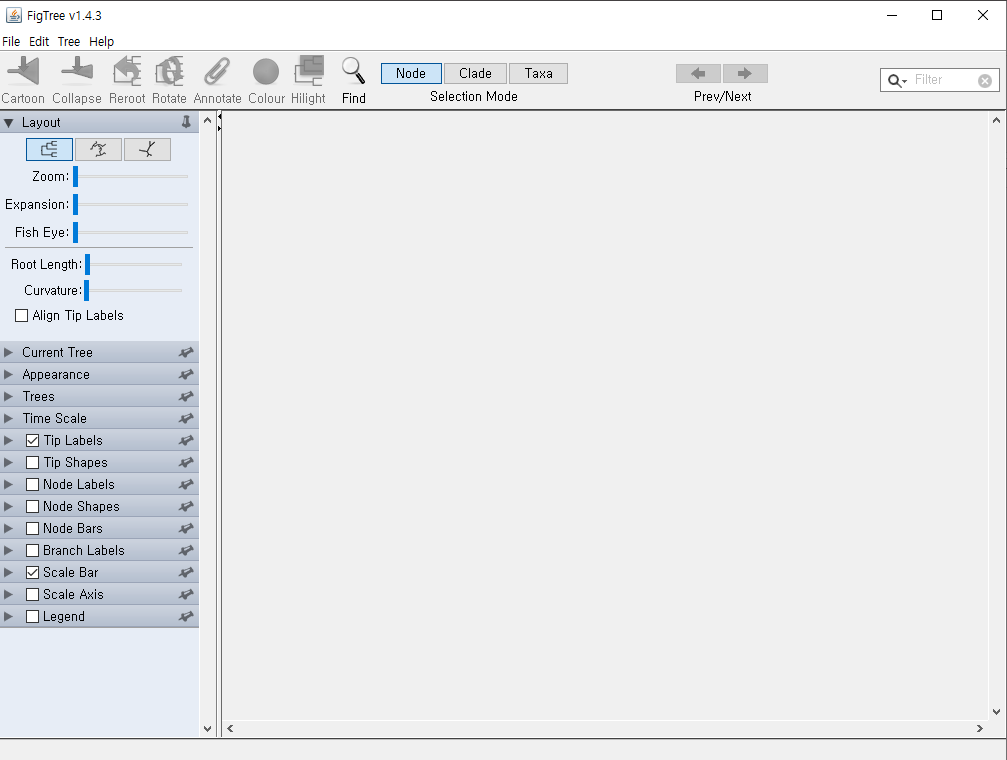
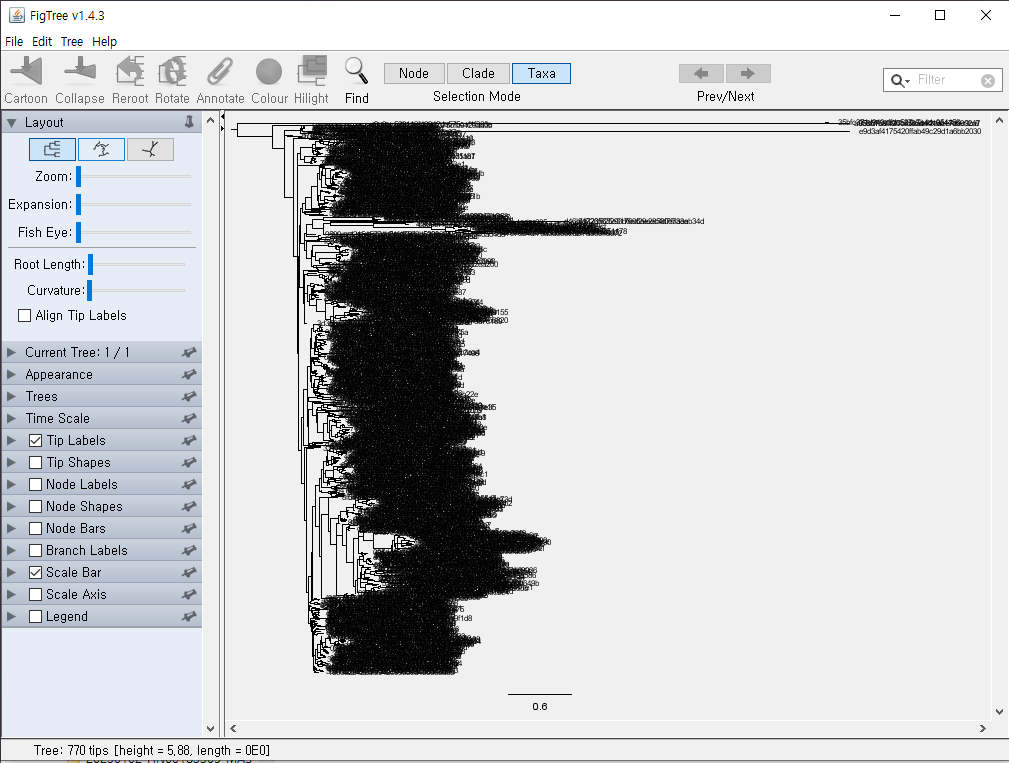
5. Open → ps_tree.tree 선택 → node의 라벨을 적으라는 알림 창에 "OTU" 입력
4. Phylotree.js [5]
이는 웹페이지에서 사용 가능하도록 제작된 javascript를 배이스로 한 tree의 시각화 라이브러리이다. 이 라이브러리는 Newick 계통수 형식을 input파일로 받는다. 계통수는 linear(기본 덴드로그램)한 구조와 Radial(원형)만 사용 가능하다. 온라인으로 편하게 그릴 수 있는 것이 장점이다. 한마디로 심플하고 깔끔하다.
- 웹페이지 : https://phylotree.hyphy.org/
Phylotree.js 에서 계통수 보기
1. 홈페이지 접속
2. 파일 선택하기
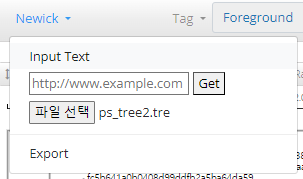
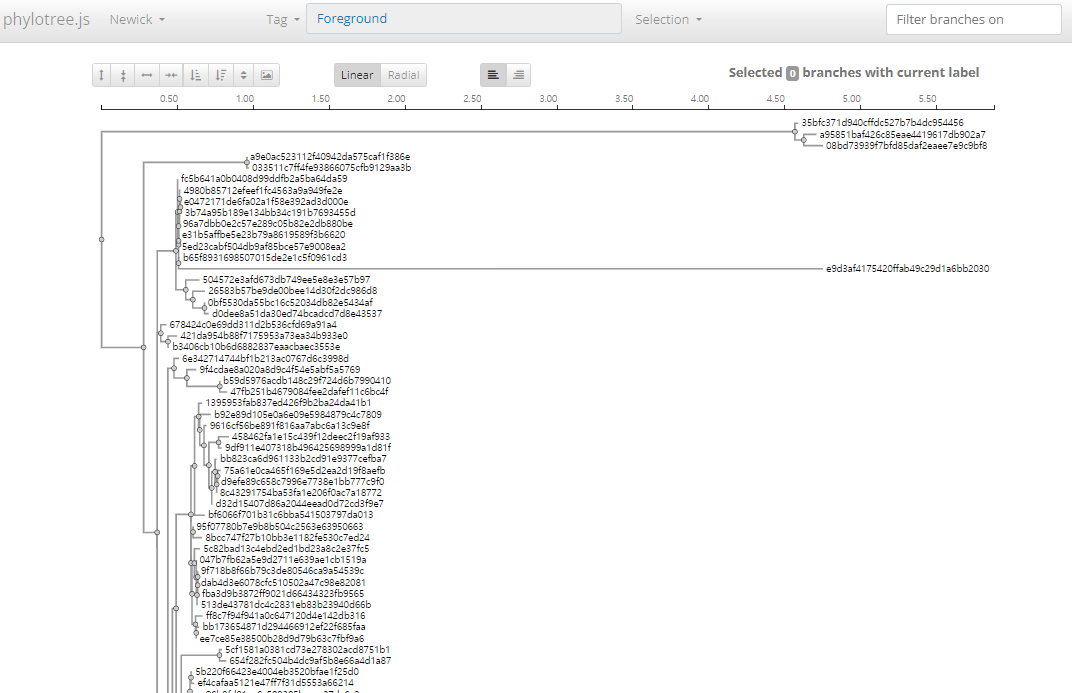
3. 계통수 보기
5. ggtree [7]
ggtree는 R환경에서 구동하는 패키지로 계통수의 시각화에 사용된다. 또한 계통수 데이터를 data frame구도로 쉽게 바꿀 수 있으며, 여러 계통수 형식을 input 파일로 사용한다. 또한 미생물 데이터 분석에 사용되는 phyloseq 패키지의 데이터를 input으로도 사용한다. 👍
- 공식 홈페이지 : https://guangchuangyu.github.io/software/ggtree/
- 관련 튜토리얼 : 블로그링크
ggtree에서 계통수 보기
1. 패키지 설치
if (!require("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("ggtree")2. tree파일 읽어오기
library(ggtree)
tr <- read.newick("./ps_tree2.tre")2. 시각화
ggtree(tr, layout="circular") + geom_tiplab(size = 1)
ggtree는 화려한 시각화를 하기 좋지만, R언어를 사용해야 하는 부담이 있다.
| References
- 계통수 시각화 프로그램 리스트 : https://en.wikipedia.org/wiki/List_of_phylogenetic_tree_visualization_software
- 원문 안의 출처
- Kumar, S., Stecher, G., & Tamura, K. (2016). MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular biology and evolution, 33(7), 1870-1874.
- Huson, D. H., & Scornavacca, C. (2012). Dendroscope 3: an interactive tool for rooted phylogenetic trees and networks. Systematic biology, 61(6), 1061-1067.
- http://tree.bio.ed.ac.uk/software/figtree/
- http://tree.bio.ed.ac.uk/software/beast/
- Shank, S. D., Weaver, S., & Pond, S. L. K. (2018). phylotree. js-a JavaScript library for application development and interactive data visualization in phylogenetics. BMC bioinformatics, 19(1), 276.
- Delport, W., Poon, A. F., Frost, S. D., & Kosakovsky Pond, S. L. (2010). Datamonkey 2010: a suite of phylogenetic analysis tools for evolutionary biology. Bioinformatics, 26(19), 2455-2457.
- Yu, G., Smith, D. K., Zhu, H., Guan, Y., & Lam, T. T. Y. (2017). ggtree: an R package for visualization and annotation of phylogenetic trees with their covariates and other associated data. Methods in Ecology and Evolution, 8(1), 28-36.
- Perrière, G. and Gouy, M. (1996) WWW-Query: An on-line retrieval system for biological sequence banks. Biochimie, 78, 364-369.
- Stöver B C, Müller K F: TreeGraph 2: Combining and visualizing evidence from different phylogenetic analyses. BMC Bioinformatics 2010, 11:7
