
이전 글에서 생명정보학을 시작하며 알아야 할 기본 원칙들(Agile, FAIR)에 대해 이야기했습니다. 이번에는 "실제 프로젝트를 어떻게 정리하고 관리해야 할까?"라는 주제로 작성해 보았습니다.
특히, 대학원에 와서 컴퓨터 언어를 처음 배우는 생명과학 전공자들에게 프로젝트 진행에 필요한 코드 리뷰와 문서화, 폴더 구조 등에 대한 기준과 실제 예시 등을 제시해 보았습니다. 이 파트는 논문 Implementing code review in the scientific workflow: Insights from ecology and evolutionary biology에서 많은 부분을 발췌하였습니다.
1. 왜 정리가 필요할까? - 코드 리뷰의 필요성
1) 코드리뷰란?
"코드 리뷰"라는 말은 뭔가 엄청 귀찮을 일처럼 느껴질 수 있는데, 평소에도 미팅처럼 코드를 함께 읽고 실행해 보며 잘 짜였는지, 재현 가능한지를 확인하는 과정입니다.
2) 코드 리뷰에서는 어떤 것을 평가해야 하는가?
(1) 보고된 내용과 동일한 결과를 산출하는지?
- 논문이나 포스터 발표자료에 적힌 분석 방법과 실제 코드가 일치하는지?
> 사용된 패키지, 버전, 함수명까지 명시
> e.g. python의 setuptools, R의 citation() 활용
- 중요한 건 Docker 같은 컨테이너화 도구를 이용해서 분석 환경 자체를 고정해 두는 것!
(2) 코드가 실행되는지?
- 생각보다 많은 논문이 제시하는 코드는 그대로 실행되지 않습니다 (필자 경험 상 5개 중 하나꼴로 실행됨😓)
> 특히 경로 문제, 버전 문제, 혹은 내부 함수가 누락되어 있는 경우가 많습니다.
> 실행 시간이 길거나 데이터가 너무 클 경우, 작은 예제 데이터셋으로 대체해서 결과 예시를 보여주는 게 중요합니다. (실제 데이터를 못 공유하는 경우 더더욱!)
(3) 코드가 신뢰 가능한지?
- R의 testthat, Python의 pytest를 통해 오류를 확인하는 것이 좋습니다.
- 또한 중간 산출물이 예상과 맞는지를 확인하는 방법도 매우 중요합니다.
> R의 identical(), Python의 numpy.array_equal() 등을 이용해서 중간에 생성된 객체가 이전과 동일한지 체크해 봅시다.
(4) 재현 가능한지?
- 모든 분석에 랜덤성이 개입될 수 있습니다. 특히 random forest, subsampling 등이 포함된 경우 set.seed() 혹은 random.seed()는 필수입니다.
- seed만 설정하고 끝내지 말고, 경우에 따라 중요한 중간 결과는. rds 혹은. csv로 저장해 두는 것도 좋습니다.
2. 어디서부터 시작할까? - 폴더 구조/ 파이프라인 관리/ 문서화
1) 효과적인 코드리뷰를 위한 프로젝트 조직화
위에 있는 항목이 당장 실행하기 어렵다면 프로젝트 폴더부터 시작하는 것을 추천드립니다. 대표적인 모범사례를 참고해 봅시다.
A quick guide to organizing computational biology projects ( Noble WS. 2009)
(1) 프로젝트 조직화
| 폴더 구조

이 논문에서 제안하는 방식은 매우 간단합니다.
- data/: 원시 데이터, 전처리된 데이터 등
- results/: 분석 결과물
- doc/: 논문 초안, 발표자료, README 등
| 폴더 이름
추가로 결과물은 날짜별 + 분석 목적에 따라 폴더를 구분하는 것도 좋습니다. (예: 2024-06-27_result, 2024-06-27_plot 등)
혹은 정렬을 위해 01_Rawdata, 02_Doc, 03_Result처럼 순번 구조도 추천하고 있습니다.
분석 주제가 명확하게 나뉘지 않는다면 "MECE(Mutually Exclusive, Collectively Exhaustive)" 원칙도 참고하는 것도 좋습니다.
| cookiecutter
프로젝트를 여러 번 생성하는 경우 효과적인 방법은 바로 cookiecutter를 사용하는 것입니다.

또한 쿠키커더를 사용해서 원하는 템플릿을 형성하여 여러 프로젝트에 따라 동일한 폴더 구조를 형성할 수 있습니다.
- 홈페이지: https://pypi.org/project/cookiecutter
쿠키커터는 여러 언어를 지원하고, 여러 목적 (소프트웨어 배포, 웹사이트, 실험 프로젝트) 등에 대한 템플릿구조를 형성해 줍니다.
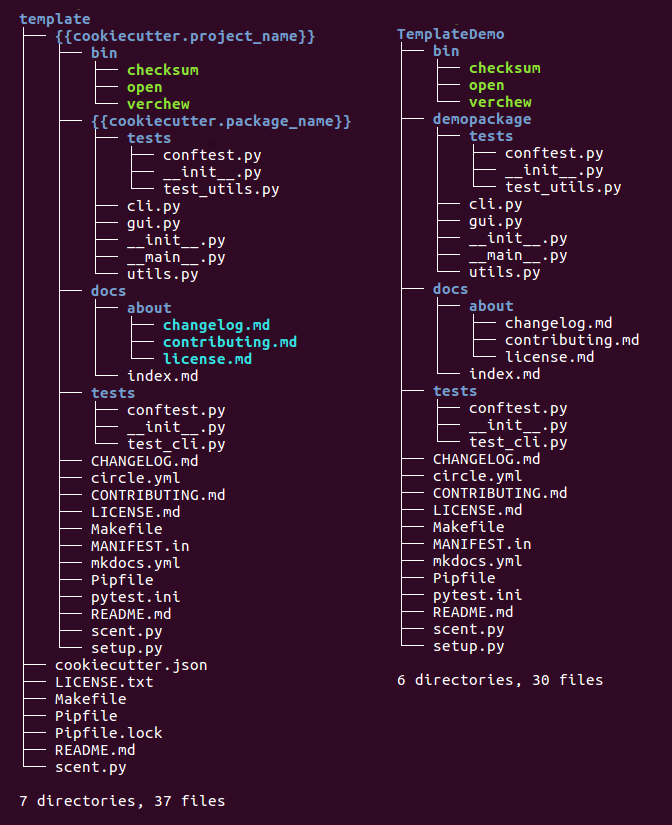
강추!
2) 폴더 및 파일 이름 규칙
폴더 구조 다음으로 중요한 것은 파일의 이름입니다. 만약 버전에 따라 모든 파일의 이름을 동일하게 만든다면, 파일을 전달받는 사람 입장에서 이게 최근 파일인지 아닌지 헷갈릴 수 있습니다.
실제로 미팅시간에 교수님도 지적하시는 부분이기도 했습니다.
연구실 내의 파일 이름 형식을 지정해 보았는데, 이는 이는 대한민국 정책 브리핑 홈페이지를 참고하였습니다.
(https://m.korea.kr/news/policyNewsView.do?newsId=148906484)
폴더 정리할 때 알아두면 좋은 꿀팁!
/폴더 정리할 때 알아두면 좋은 꿀팁!폴더 정리할 때 알아두면 좋은 꿀팁!폴더 정리할 때 알아두면 좋은 꿀팁!폴더 정리할 때 알아두면 좋은 꿀팁!폴더 정리할 때 알아두면 좋은 꿀팁 - 정책브리
www.korea.kr
| 💡 파일 이름 예시
구조는 "날짜_프로젝트_세부명_작성자_버전"입니다.
# AT 프로젝트의 진균 분석, 작성자 KSY, 버전 3
250402_AT_FungiAnalysis_ksy_v3.pptx
# AT 프로젝트의 진균 분석, 작성자 KSY, 파이널 파일
250422_AT_FungiAnalysis_ksy_f.pptx
3) 파이프라인 관리
또한 파이프라인 관리를 위해 R에서는 targets 패키지를 추천한다. 이는 아래와 같이 각 분석 단계를
| targets
library(targets)
# 데이터 처리 및 분석을 위한 타깃 정의
tar_option_set(packages = c("dplyr", "ggplot2", "phyloseq"))
# 타깃 목록
list(
tar_target(raw_data, readRDS("raw_microbiome_data.rds")), # 원시 데이터 읽기
tar_target(clean_data, clean_microbiome_data(raw_data)), # 데이터 전처리
tar_target(otu_table, generate_otu_table(clean_data)), # OTU 테이블 생성
tar_target(taxonomy, assign_taxonomy(otu_table)), # 분류학 할당
tar_target(diversity, calculate_diversity(otu_table)), # 다양성 분석
tar_target(plot, plot_diversity(diversity)) # 결과 시각화
)위 내용을 한 줄로 실행하면 아래와 같다.
library(targets)
tar_make()
하지만 dependency가 있기 때문에 수동 스크립트와 완전히 동일하진 않다. 디버깅할 때는 오히려 불편할 수도 있음. (그래서 테스트 데이터로 먼저 시도해 보는 게 좋다)
4) 메타데이터와 문서화
Github 볼 때마다 README를 보실 텐데, 어떤 패키지는 상세하게 적혀있고, 어떤 패키지는 진짜 소개글만 적혀있는 경우가 있습니다. README는 데이터를 처음 보는 사람의 이해를 위한 문서입니다. 중요한 내용은 상세하게 기술해야 합니다. 또한 README는 다음과 같은 내용이 포함되어야 합니다. 또한 작성은 FAIR원칙을 따르는 것이 좋습니다.
- 프로젝트 개요
- 작성자, 데이터 소유자
- 프로젝트 목표
- 사용된 언어/ 패키지
- 데이터의 기술 (출처, 형식, 위치)
- 코드 라이선스
소프트웨어를 위한 자세한 문서 작성은 QuickStart guide와 --help 문서 등의 기술도 포함된다. 자세한 사항은 아래 논문을 읽어보면 좋습니다!

3. 조금 더 완벽한 코드를 위해서
1) 코드 가독성
가독성을 높이는 기본 원칙은 다음과 같다.
a. 반복적인 기능은 함수화 하자
b. 절대경로가 아닌 상대경로로 설정하자 (협업에 중요합니다)
c. 코드에 주석을 단계별로 명확히 작성하자
d. - 변수 이름을 명확하게 작성하자.
- e.g. data 가 아니라 otu_table, res 대신 alpha_results
가독성을 높여주는 도구는 다음과 같다.
- R에서 styler 패키지: 불필요한 공백제거 +정렬 적용
- Python의 pycodestyle 패키지 : PEP8 스타일로 정리
2) 출력 재현성
Monte Carlo처럼 랜덤 요소가 많은 경우, seed 고정만으로는 부족합니다. 중간 결과를. rds,. csv,. feather 등으로 저장해 두고 불러오는 방식이 훨씬 안전합니다.
| 출처
- Ivimey-Cook, E. R., Pick, J. L., Bairos-Novak, K. R., Culina, A., Gould, E., Grainger, M., Marshall, B. M., Moreau, D., Paquet, M., Royauté, R., Sánchez-Tójar, A., Silva, I., & Windecker, S. M. (2023). Implementing code review in the scientific workflow: Insights from ecology and evolutionary biology. Journal of Evolutionary Biology, 36(10), 1347-1356. https://doi.org/10.1111/jeb.14230
- Noble W. S. (2009). A quick guide to organizing computational biology projects. PLoS computational biology, 5(7), e1000424. https://doi.org/10.1371/journal.pcbi.1000424
- Lee B. D. (2018). Ten simple rules for documenting scientific software. PLoS computational biology, 14(12), e1006561. https://doi.org/10.1371/journal.pcbi.1006561
4. 연구실에서 적용해 가야 할 사항은?
실험실 차원에서 코드 정리와 문서화 문화를 정착시키려면, 현재 프로젝트의 상태와 구성원의 역할에 따라 단계적으로 접근하는 것이 중요합니다. 아래는 프로젝트 상태별로 실천할 수 있는 방안들입니다.
1) 종료된 프로젝트의 경우
- 코드 리뷰: 분석 코드가 논문/발표 결과와 일치하는지 확인
- GitHub 업로드: 프로젝트 구조를 정리해 GitHub에 업로드
- README 및 실행 가이드 작성: 외부인 또는 랩 구성원이 다시 봤을 때 분석 흐름을 따라갈 수 있도록 작성 =
2) 진행 중인 프로젝트의 경우
아직 진행 중인 프로젝트라면 지금이라도 정리 습관을 들이는 것이 중요합니다
- 폴더 구조 명확히 설정: 예) 01_rawdata, 02_script, 03_result, README.md 등
- 버전 관리 도입: GitHub 또는 GitLab으로 버전 관리 시작
- 실수 복구, 협업 이력 추적에 유용
- 작업 단위별 커밋 습관: 코드 변경 내역을 남겨야 실험/분석 과정을 되짚을 수 있음
3) 교육자료
- 만약 분석 파이프라인에 대해 설명 자료가 있다면, Jupyter Note나 Rmarkdown을 사용하자
- 또한 공유를 위해 구글 드라이브, Notion 등 공유 가능한 문서로 정리
- 문서 작성자는 교육 자료 내의 버전, 작성날짜, 작성자를 명시해야 한다
4) 분석 파이프 라인
- 반복 분석은 Nextflow, Snakemake 등을 활용해 자동화
- 시각화는 R Shiny를 사용한 Dashboard 사용을 고려
🙋♀️졸업 전에 실험실 시스템을 한번 구축해 보는 걸 추천합니다. 여러분의 코드 정리 습관이 후배들에게 그대로 전달됩니다.
또한 상세한 문서 기술은 제 자산이 될 수 있고, 졸업하고 나서도 랩이 발전하는 길이기도 합니다!
5. 그러나.. 할 시간은 있고?
저도 notion을 통해서 제가 배우고 한 일들을 모두 기입하고 있는데요.. 가끔 적으면서 의미가 있을까? 하는 생각도 듭니다.
관련 고민을 또 서치 하다가 공감되는 레딧 게시글을 가져왔습니다. 사실 이런 질문을 보거나 공유할 곳이 레딧이 유일한 것 같습니다.
From the bioinformatics community on Reddit
Explore this post and more from the bioinformatics community
www.reddit.com
위 글에서 여러 댓글을 인용하면 다음과 같습니다.
- "전문성 이전에 중요한 것은 그것들을 배우고 실천할 수 있는 시간이다 (하지만 대학원생은 시간이 없지..)."
- "네가 프로젝트 및 코드 유지 관리에 신경을 쓴다고 해도 졸업하고 얻을 수 있는 것이 있을까? 결국 기여하는 것은 랩 단위가 아니라 더 큰 커뮤니티 단위에 기여하는 것이 좋을 것이다 (Galaxy, Biobonductor 등)."
- "학계는 이것에 관심이 없다. 재현성 계획 및 효과적인 문서화와 파이프라인은 출판에 아무런 도움이 되지 않는다. "
- "출판 이후에 아무도 그것에 관심을 가지지 않고 유지관리 (git hub commit)조차 하지 않는다."
등..
정말 맞는 말이죠??😗
사실 이 글에 적힌 말들이 모두 다 알고있는 내용이라고 생각합니다. 그러나 이를 연구책임자나 과제관리처에서 딱히 신경 쓰지 않고 있을 겁니다. 논문을 게재할 때에도 탑급 저널이 아니면 크게 장려하고 있지 않습니다.
그러나 내가 스스로 정리한 코드와 문서는 언젠가 미래의 나 & 연구실 후배 & 분야의 후배에게 도움이 될 것이라고 믿습니다.
완벽하지 않아도 괜찮으니, 조금씩 시작해 보는 것이 어떨까요?
| 이전 편
생물정보학에서 살아남기 01: 소프트웨어 개발 원칙과 Agile, FAIR
🙋♀️안녕하세요. 김해김 씨 99대손입니다.오늘은 생명정보학을 공부하는 분들, 특히 컴퓨터를 대학원에 와서 익히게 된 생명과학 전공자들에게 꼭 들려주고 싶은 이야기를 정리해 보았습
bio-kcs.tistory.com
| 다음 편 예고
>> 생물정보학에서 살아남기 03: GitHub 실전 가이드
읽어주셔서 감사합니다!
