
| 준비물
- 설치된 BLASTn (참고 : blast 설치)
- fas/fasta파일
fas/fasta파일은 서열과 서열의 아이디로 구성되어 있으며, 아이디는 > 다음에 오는 글자를 말한다.
| 자동화 함수 만들기
1. 빈 문서 만들기
vi blast_script.sh
2. 생성된 문서에 아래 내용 붙여 넣기
- 만약 fasta서열이면, for file in *.fasta; do 로 바꾸기
#!/bin/bash
mkdir ./results/
output_dir="./results/"
for file in *.fas; do
output_file="${output_dir}${file%.*}.csv"
output_file_sum="${output_dir}${file%.*}_sum.csv"
blastn -db ~/Reference/blastdb/16S_ribosomal_RNA \
-query "${file}" \
-task blastn \
-dust no \
-outfmt "7 delim=, qacc sacc evalue bitscore qcovus pident sscinames" \
-max_target_seqs 5 > "$output_file"
grep -v '#' "$output_file" > "$output_file_sum"
doneblast결과를 Top 5만 저장하겠다는 스크립트 이다.
3. 스크립트 파일에 실행 권한 부여하기
chmod +x blast_script.sh
4. 스크립트 실행하기
./blast_script.sh
| 결과 확인하기
cd results/
ll생성된 두 개의 파일을 볼 수 있다.

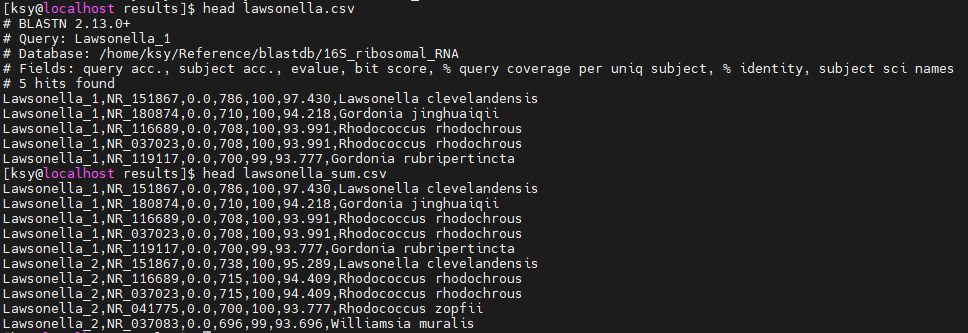
*.csv파일은 blast의 전체 결과를 표시한 것이고,
*_sum.csv파일은 *.csv파일에서 주석 처리된 부분을 제외하고 저장된 문서이다.
| +) Top 5와 Top1결과 저장하는 스크립트
#!/bin/bash
mkdir ./results/
output_dir="./results/"
for file in *.fas; do
output_file="${output_dir}${file%.*}.csv"
output_file_sum="${output_dir}${file%.*}_sum.csv"
blastn -db ~/Reference/blastdb/16S_ribosomal_RNA \
-query "${file}" \
-task blastn \
-dust no \
-outfmt "7 delim=, qacc sacc evalue bitscore qcovus pident sscinames" \
-max_target_seqs 5 > "$output_file"
grep -v '#' "$output_file" > "$output_file_sum"
output_file_t1="${output_dir}${file%.*}_t1.csv"
output_file_t1_sum="${output_dir}${file%.*}_t1_sum.csv"
blastn -db ~/Reference/blastdb/16S_ribosomal_RNA \
-query "${file}" \
-task blastn \
-dust no \
-outfmt "7 delim=, qacc sacc evalue bitscore qcovus pident sscinames" \
-max_target_seqs 1 > "$output_file_t1"
grep -v '#' "$output_file_t1" > "$output_file_t1_sum"
done반응형
