
| 개요
1세대인 Sanger sequencing의 정확도는 매우 높지만, 한 번의 한 서열만 읽을 수 있다는 단점이 있다. 이를 극복한 것이 서열을 대량으로 읽어내리는 Massive parellel sequencing(MPS)이다. 이는 NGS sequencing 혹은 2세대 시퀀싱 기술이라고 불린다.
2세대 시퀀싱 기술은 크게 두 범주로 나뉜다.
1. Sequencing by hybridization
1980년대에 고안되었으며, 이미 알고 있는 서열과 알고자 하는 서열의 binding을 통해 기존의 서열과 일치 여부를 확인한다. 크게 두 방식으로 분류 가능한데, 하나는 알고자 하는 target sequence를 셀에 고정하고, 서열 조각을 binding 하여 binding efficiency의 차이로 sequencing을 해 나가는 방식과, immobilized nucleotide probe에 sequencing target을 붙이는 방식이다.
2. Sequencing by hybridization(SBS)
SBS는 2000년도 후반에 개발 되었으며, solid support나 wells 안에서 시퀀싱이 실행된다. Sanger의 기술처럼 ddNTP를 사용하지 않지만, 합성된 염기를 측정하기 위해(합성될 때 방출되는 형광 분자를 감지하기 위해)를 위해 일부 합성을 지연하는 과정이 사용된다.
SBS의 sequencing 과정을 간략하게 살펴보자.
1. Library preparation (DNA library 구축) : PCR을 통해 여러 clone 서열들을 만들어 낸다.
2. Sequenced by synthesis : 만들어진 clone을 주형가닥 삼아 완전한 상보적인 서열을 만들어 낸다. (Sanger 시퀀싱 방법인 chain termination을 사용하지 않는다.)
3. Sequencing : 증폭된 DNA template를 동시에 대량으로(massively parallel) 읽어나간다.
| illumina sequencing platform
Sanger sequencing과 다른 2세대 시퀀싱 기술의 단점은 sequencing이 진행될수록 후반부 서열의 퀄리티가 떨어지는 것이다. illumina사는 이를 보완하기 위해 서열의 forward, reverse를 각각 읽어 들여서 서열의 퀄리티를 높이는 방법을 선택하였다.
이 방법은 캠브릿지 대학의 Shankar Balasubramanian와 David Klenerman이 고안했다. 이들은 Solexa사를 창업하였으며, 이후 회사는 illumina에 인수되었다.
| illumina sequencing 과정
1. Genomic library 제작
DNA가 정제된 후, 시퀀싱에 사용할 DNA library가 만들어 진다. 일루미나에 사용되는 library는 100~500bp의 길이로 잘려진 DNA 조각들이다. 이러한 DNA library를 만드는 방법은 두 종류가 있다.
1) tagmentation
transposase(전이효소)를 통해 서열을 50~500bp사이로 잘라내며 adapter를 동시에 추가한다.
2) sonification(초음파)
초음파를 사용하여 DNA를 조각으로 절단한다. 이후, 양쪽의 adapter는 T7 DNA Polymerase와 T4 DNA ligase를 이용해서 연결한다.
2. 서열에 Adapter 붙이기
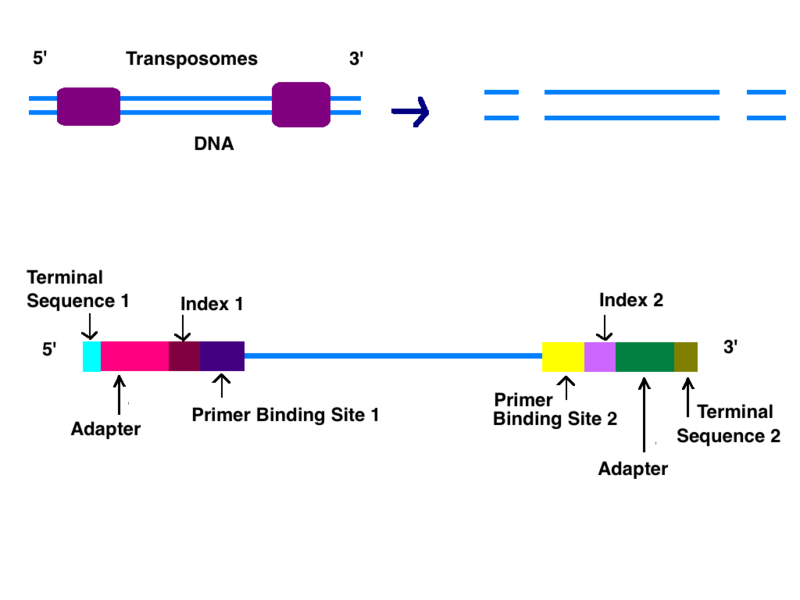
서열에 추가되는 adapter는 총 3부분으로 이루어져 있다.
1) barcode sequence (indices)
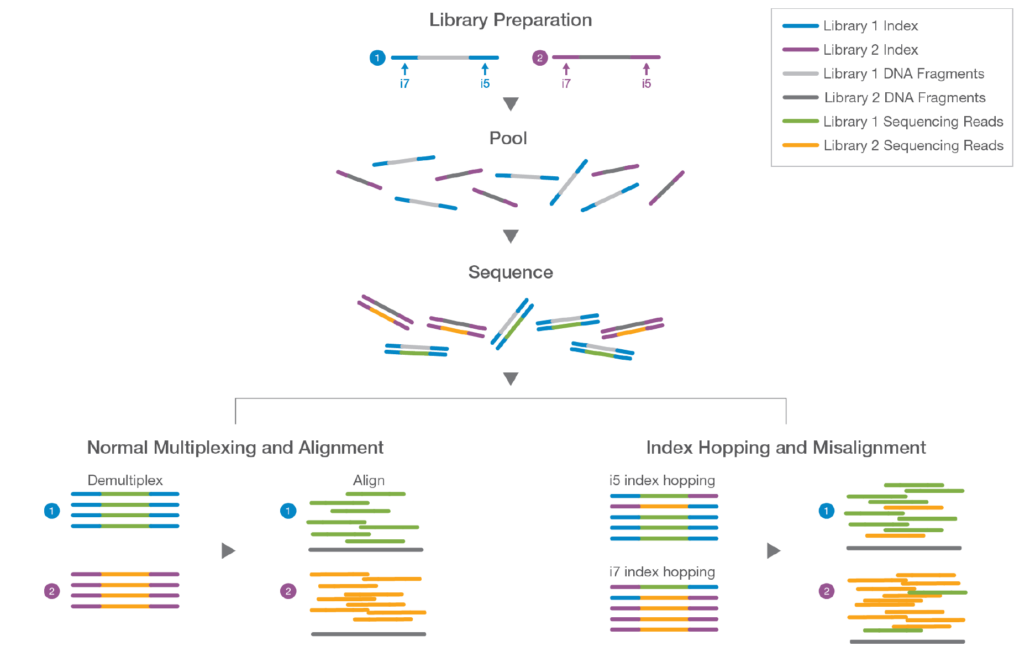
- 각 서열이 어떤 정보를 담고 있는지 표시해 주는 서열이다. 일반적으로 6 base pair 이상의 길이가 사용되며, 이를 이용해 최대 96개의 샘플을 구분할 수 있다. 즉 한번 시퀀싱을 돌릴 때, 최대 96개의 샘플을 동시에 돌릴 수 다는 뜻이다.
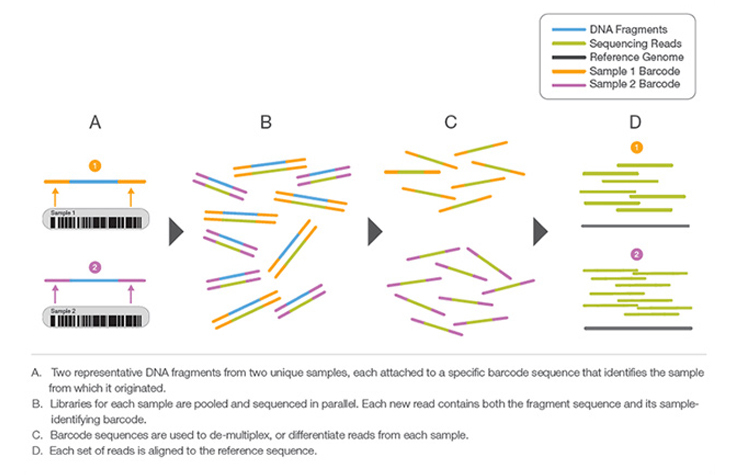
- 이처럼 바코드를 통해 서열을 구분하지만 같은 시퀀싱 Run을 돌리는 것을 Multiplexing이라고 하며, 분석 단계에서 바코드 서열을 통해 각 샘플에 해당하는 서열을 나누는 단계를 Demultiplexing이라고 한다. 아래 그림은 Demultiplexing 단계를 나타낸다.
2) sequence complementary to solid support
- 시퀀싱이 이루어지는 고체 부분에 부착되기 위한 서열을 말한다. DNA조각들은 acrylamide로 코팅된 glass flow cell에서 합성이 진행되는데, 이때 서열을 cell의 바닥에 존재하는 oligonucleotides(짧은 서열 조각 = solid support)에 고정된다.

3) sequencing primer
- DNA를 증폭하기 위해 사용된다.
3. Bridge amplification and clonal amplification
DNA library가 증폭되는 과정은 아래 그림과 같다. 그림에 나타난 순서를 하나하나 알아보자.
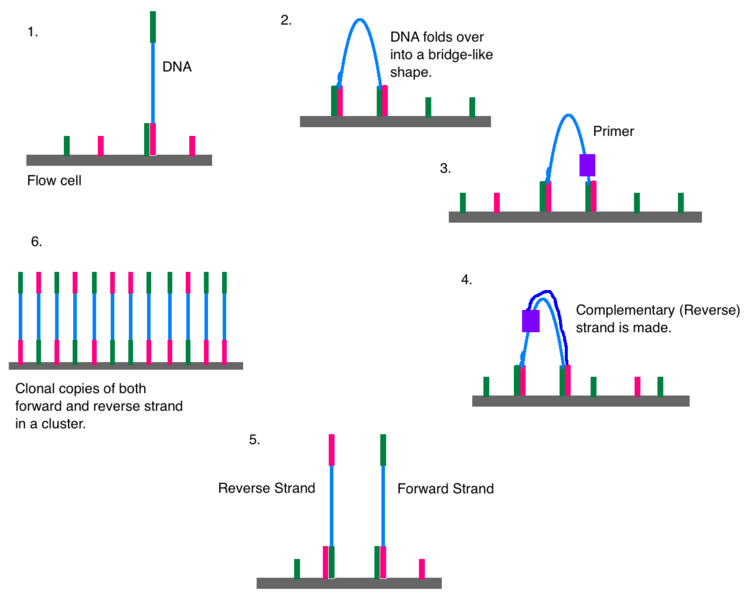
1) glass flow cell의 고정된 서열(support)에 DNA library의서열이 부착된다.
2) 서열의 다른 한 끝쪽이 또 다른 support와 연결되어 brigde형태가 형성된다.
3) DNA polymerase가 brigde 구조를 가진 서열의 primer를 인식하고, 상보적인 reverse서열을 만든다. 합성이 완료된 double stranded DNA는 분리된다.
4-5) 결과적으로, forward와 reverse strand가 완성된다.
6) 2-3-4과정이 반복되어 같은 DNA를 가진 forward, reverse서열이 집단(cluster)을 이루게 된다.
4. Sequence by synthesis
- clonal amplification가 끝나면, 먼저 forward strand를 인식하기 위해 모든 reverse 서열은 씻겨낸다. 이후 오직 forward strand만 남게된다. 남은 서열에서 형광분자가 태그 된 dNTP를 사용해서 상보적인 서열이 합성된다.
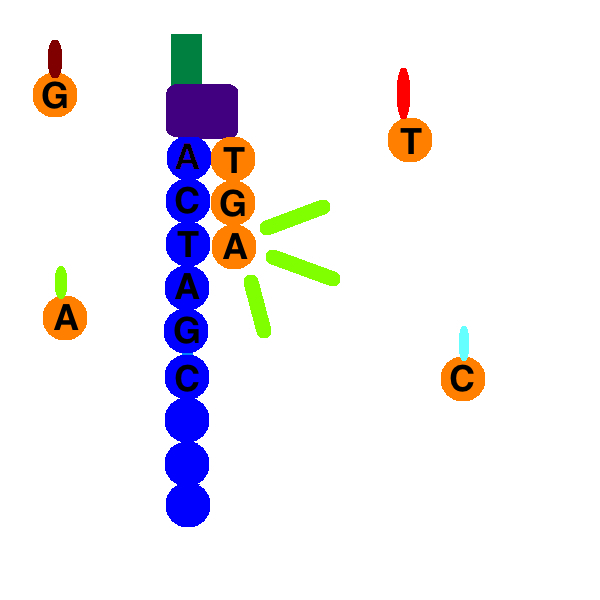
- 하나의 합성 주기마다 하나의 base만 합성된다. 이는 Fluorophore(합성물질)이 다른 염기가 합성되는 것을 막기때문이다. dNTP가 합성되면 각 염기마다 다른 색의 빛을 방출한다. 이때 측정 기계는 방출되는 빛을 감지한다. 이후, fluorophore와 dNTP는 씻겨져 내려가고, 다시 한번 이 과정을 반복한다.
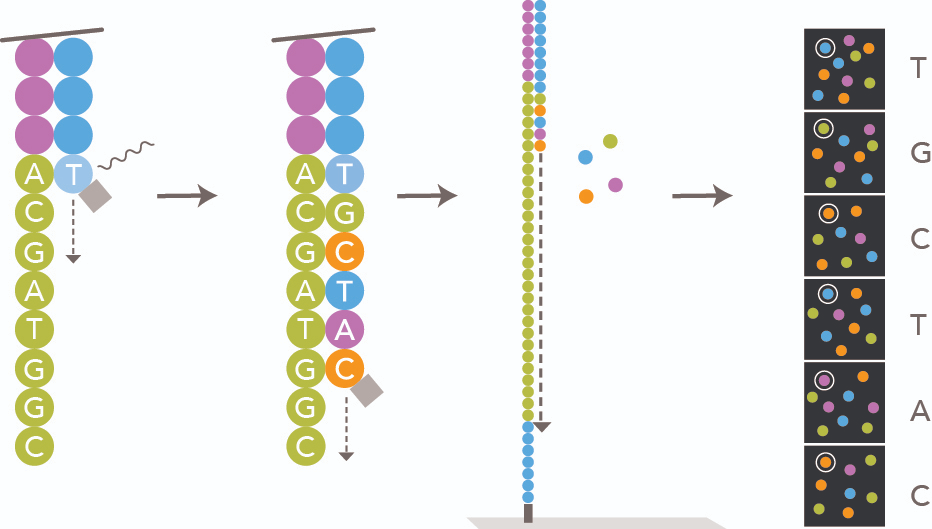
- 이때 같은 DNA 주형을 가지는 서열에서 합성되는 염기가 모두 같은 종류의 염기일 경우 퀄리티가 높고, 염기의 종류가 다양할 수록 퀄리티는 떨어진다.
5. Data Analysis
- 한 번 시퀀싱 할 때(서열을 읽어 들일 때), 수 백만개의 서열을 읽어 들이며, 한 서열을 1000개 이상의 복사본을 가지게 된다. 읽힌 서열을 forward와 reverse에서 겹치는(overapping)부분을 기준으로 서열을 합쳐(dada2의 merge단계)서 완전한 서열 조각이 완성된 후 분석된다.
| illumina sequencing 의 단점
- illumina사의 시퀀싱은 긴 길이의 서열을 읽어 들이지 못하고, reference가 존재하지 않는 서열의 경우에는 조립의 어려움을 겪는다.
- 또한 중합효소의 부정확한 서열 합성, index hopping(=index switching 잘못된 서열에 부착되는 것), 키메라 서열의 생산 및 PCR 단계의 오류 등이 생길 수 있다.
| 용어
Chimera sequence
- PCR과정에서 생긴 서열을 말하며, True서열이 섞여서 생긴 가짜 서열을 말한다.

| 출처
- 정규열, 신지원, 차세대 염기 분석기술(Next-generation Sequencing Technologies), BT News-신기술
- https://en.wikipedia.org/wiki/Massive_parallel_sequencing
- https://en.wikipedia.org/wiki/Illumina_dye_sequencing
- Slatko BE, Gardner AF, Ausubel FM. Overview of Next-Generation Sequencing Technologies. Curr Protoc Mol Biol. 2018 Apr;122(1):e59. doi: 10.1002/cpmb.59. PMID: 29851291; PMCID: PMC6020069.

| 개요
1세대인 Sanger sequencing의 정확도는 매우 높지만, 한 번의 한 서열만 읽을 수 있다는 단점이 있다. 이를 극복한 것이 서열을 대량으로 읽어내리는 Massive parellel sequencing(MPS)이다. 이는 NGS sequencing 혹은 2세대 시퀀싱 기술이라고 불린다.
2세대 시퀀싱 기술은 크게 두 범주로 나뉜다.
1. Sequencing by hybridization
1980년대에 고안되었으며, 이미 알고 있는 서열과 알고자 하는 서열의 binding을 통해 기존의 서열과 일치 여부를 확인한다. 크게 두 방식으로 분류 가능한데, 하나는 알고자 하는 target sequence를 셀에 고정하고, 서열 조각을 binding 하여 binding efficiency의 차이로 sequencing을 해 나가는 방식과, immobilized nucleotide probe에 sequencing target을 붙이는 방식이다.
2. Sequencing by hybridization(SBS)
SBS는 2000년도 후반에 개발 되었으며, solid support나 wells 안에서 시퀀싱이 실행된다. Sanger의 기술처럼 ddNTP를 사용하지 않지만, 합성된 염기를 측정하기 위해(합성될 때 방출되는 형광 분자를 감지하기 위해)를 위해 일부 합성을 지연하는 과정이 사용된다.
SBS의 sequencing 과정을 간략하게 살펴보자.
1. Library preparation (DNA library 구축) : PCR을 통해 여러 clone 서열들을 만들어 낸다.
2. Sequenced by synthesis : 만들어진 clone을 주형가닥 삼아 완전한 상보적인 서열을 만들어 낸다. (Sanger 시퀀싱 방법인 chain termination을 사용하지 않는다.)
3. Sequencing : 증폭된 DNA template를 동시에 대량으로(massively parallel) 읽어나간다.
| illumina sequencing platform
Sanger sequencing과 다른 2세대 시퀀싱 기술의 단점은 sequencing이 진행될수록 후반부 서열의 퀄리티가 떨어지는 것이다. illumina사는 이를 보완하기 위해 서열의 forward, reverse를 각각 읽어 들여서 서열의 퀄리티를 높이는 방법을 선택하였다.
이 방법은 캠브릿지 대학의 Shankar Balasubramanian와 David Klenerman이 고안했다. 이들은 Solexa사를 창업하였으며, 이후 회사는 illumina에 인수되었다.
| illumina sequencing 과정
1. Genomic library 제작
DNA가 정제된 후, 시퀀싱에 사용할 DNA library가 만들어 진다. 일루미나에 사용되는 library는 100~500bp의 길이로 잘려진 DNA 조각들이다. 이러한 DNA library를 만드는 방법은 두 종류가 있다.
1) tagmentation
transposase(전이효소)를 통해 서열을 50~500bp사이로 잘라내며 adapter를 동시에 추가한다.
2) sonification(초음파)
초음파를 사용하여 DNA를 조각으로 절단한다. 이후, 양쪽의 adapter는 T7 DNA Polymerase와 T4 DNA ligase를 이용해서 연결한다.
2. 서열에 Adapter 붙이기

서열에 추가되는 adapter는 총 3부분으로 이루어져 있다.
1) barcode sequence (indices)
- 각 서열이 어떤 정보를 담고 있는지 표시해 주는 서열이다. 일반적으로 6 base pair 이상의 길이가 사용되며, 이를 이용해 최대 96개의 샘플을 구분할 수 있다. 즉 한번 시퀀싱을 돌릴 때, 최대 96개의 샘플을 동시에 돌릴 수 다는 뜻이다.
- 이처럼 바코드를 통해 서열을 구분하지만 같은 시퀀싱 Run을 돌리는 것을 Multiplexing이라고 하며, 분석 단계에서 바코드 서열을 통해 각 샘플에 해당하는 서열을 나누는 단계를 Demultiplexing이라고 한다. 아래 그림은 Demultiplexing 단계를 나타낸다.

2) sequence complementary to solid support
- 시퀀싱이 이루어지는 고체 부분에 부착되기 위한 서열을 말한다. DNA조각들은 acrylamide로 코팅된 glass flow cell에서 합성이 진행되는데, 이때 서열을 cell의 바닥에 존재하는 oligonucleotides(짧은 서열 조각 = solid support)에 고정된다.

3) sequencing primer
- DNA를 증폭하기 위해 사용된다.
3. Bridge amplification and clonal amplification
DNA library가 증폭되는 과정은 아래 그림과 같다. 그림에 나타난 순서를 하나하나 알아보자.

1) glass flow cell의 고정된 서열(support)에 DNA library의서열이 부착된다.
2) 서열의 다른 한 끝쪽이 또 다른 support와 연결되어 brigde형태가 형성된다.
3) DNA polymerase가 brigde 구조를 가진 서열의 primer를 인식하고, 상보적인 reverse서열을 만든다. 합성이 완료된 double stranded DNA는 분리된다.
4-5) 결과적으로, forward와 reverse strand가 완성된다.
6) 2-3-4과정이 반복되어 같은 DNA를 가진 forward, reverse서열이 집단(cluster)을 이루게 된다.
4. Sequence by synthesis
- clonal amplification가 끝나면, 먼저 forward strand를 인식하기 위해 모든 reverse 서열은 씻겨낸다. 이후 오직 forward strand만 남게된다. 남은 서열에서 형광분자가 태그 된 dNTP를 사용해서 상보적인 서열이 합성된다.

- 하나의 합성 주기마다 하나의 base만 합성된다. 이는 Fluorophore(합성물질)이 다른 염기가 합성되는 것을 막기때문이다. dNTP가 합성되면 각 염기마다 다른 색의 빛을 방출한다. 이때 측정 기계는 방출되는 빛을 감지한다. 이후, fluorophore와 dNTP는 씻겨져 내려가고, 다시 한번 이 과정을 반복한다.

- 이때 같은 DNA 주형을 가지는 서열에서 합성되는 염기가 모두 같은 종류의 염기일 경우 퀄리티가 높고, 염기의 종류가 다양할 수록 퀄리티는 떨어진다.
5. Data Analysis
- 한 번 시퀀싱 할 때(서열을 읽어 들일 때), 수 백만개의 서열을 읽어 들이며, 한 서열을 1000개 이상의 복사본을 가지게 된다. 읽힌 서열을 forward와 reverse에서 겹치는(overapping)부분을 기준으로 서열을 합쳐(dada2의 merge단계)서 완전한 서열 조각이 완성된 후 분석된다.
| illumina sequencing 의 단점
- illumina사의 시퀀싱은 긴 길이의 서열을 읽어 들이지 못하고, reference가 존재하지 않는 서열의 경우에는 조립의 어려움을 겪는다.
- 또한 중합효소의 부정확한 서열 합성, index hopping(=index switching 잘못된 서열에 부착되는 것), 키메라 서열의 생산 및 PCR 단계의 오류 등이 생길 수 있다.

| 용어
Chimera sequence
- PCR과정에서 생긴 서열을 말하며, True서열이 섞여서 생긴 가짜 서열을 말한다.

| 출처
- 정규열, 신지원, 차세대 염기 분석기술(Next-generation Sequencing Technologies), BT News-신기술
- https://en.wikipedia.org/wiki/Massive_parallel_sequencing
- https://en.wikipedia.org/wiki/Illumina_dye_sequencing
- Slatko BE, Gardner AF, Ausubel FM. Overview of Next-Generation Sequencing Technologies. Curr Protoc Mol Biol. 2018 Apr;122(1):e59. doi: 10.1002/cpmb.59. PMID: 29851291; PMCID: PMC6020069.
