- Moving Picture data를 이용해 microbiomeMarker 로 분석을 해보려는데, clagoram이 그려지지 않았다.
- cladogram이란 lefse분석 이후 각 그룹을 대표하는 biomarker 생물들을 각 정렬하고 어떤 type에서 많이 상대적으로 나타나는지 보여주는 그림이다.
- 아래와 같이 나타남

일단 패키지 설치 하고
if (!requireNamespace("BiocManager", quietly = TRUE)) {
install.packages("BiocManager")
}
BiocManager::install("microbiomeMarker")
library(microbiomeMarker) #
Moving Picture 데이터로 만든 phyloseq [ps]를
1. 10% prevalence로 선발한 후
2. body site 를 factor로 치환
3. tongue와 gut만 subset
4. lefse분석 ㄱ ㄱ
install.packages("devtools")
library(devtools)
devtools::install_github("vmikk/metagMisc")
library(metagMisc)
# moving-picture data
# 10% prevalence로 선발
ps.10 <- phyloseq_filter_prevalence(ps, prev.trh = 0.1, abund.type = "total")
sample_data(ps.10)$body.site <- as.factor(sample_data(ps.10)$body.site) # factorize for DESeq2
ps.10.taxa <- tax_glom(ps.10, taxrank = 'Species', NArm = FALSE) # species기준으로 같은 놈이면 합쳐라
# 장과 혀 부분만 추출
ps.10.taxa.sub<-phyloseq::subset_samples(ps.10.taxa, body.site %in% c("gut","tongue"))
# lefse 분석 돌리기
lef_out_cut4 <- run_lefse(ps.10.taxa.sub, group = "body.site",
norm = "CPM", # normalization
# CPM : pre-sample normalization of the sum of the values to 1e+06
# 이미 sample normarlization했으면 "none"으로 하면 됨
taxa_rank = "Species" ,
kw_cutoff = 0.05,
wilcoxon_cutoff = 0.05,
bootstrap_n = 999,
lda_cutoff = 4) # 일단은 낮게 잡자
plot_ef_bar(lef_out_cut4)

plot_cladogram(lef_out_01_cut2, color = c("darkgreen", "red"))
# Error in if (n <= length(x)) { : missing value where TRUE/FALSE neededcladogram을 그리려고 하면 에러가 뜬다
검색해보니 NA 나 그런 데이터가 있어서 그렇다는게 아무리 검색해도 모르겠다
그래서 taxa_rank를 all로 바꾸고 다시 해 봤다
lef_out_cut4_2 <- run_lefse(ps.10.taxa.sub, group = "body.site",
norm = "CPM", # normalization
taxa_rank = "all" , # Species -> all로 변경
kw_cutoff = 0.05,
wilcoxon_cutoff = 0.05,
bootstrap_n = 999,
lda_cutoff = 4)
plot_cladogram(lef_out_cut4_2, color = c("darkgreen", "red"))
이제 된다ㅠ all로 할때만 가능한가부다....
하지만 내가 원하는건 species level에서 각 그룹마다 차이가 나는 놈들을 보고싶은건데..
그럼 원하는 species이름을 뽑아보자
lef_table <- marker_table(lef_out_01_cut2)
lef_table <- lef_table[lef_table$ef_lda >= 4.0 & lef_table$pvalue< 0.05& lef_table$padj< 0.05,1:2 ]
여기서 s__가 포함된 그룹만 추출하면 된다
이때 아주 유용한 grep()을 사용하자(excel에서 ctrl+F와 같다)
ss <- grep('s__', lef_table$feature)
lef_table_ss <- lef_table[ss,]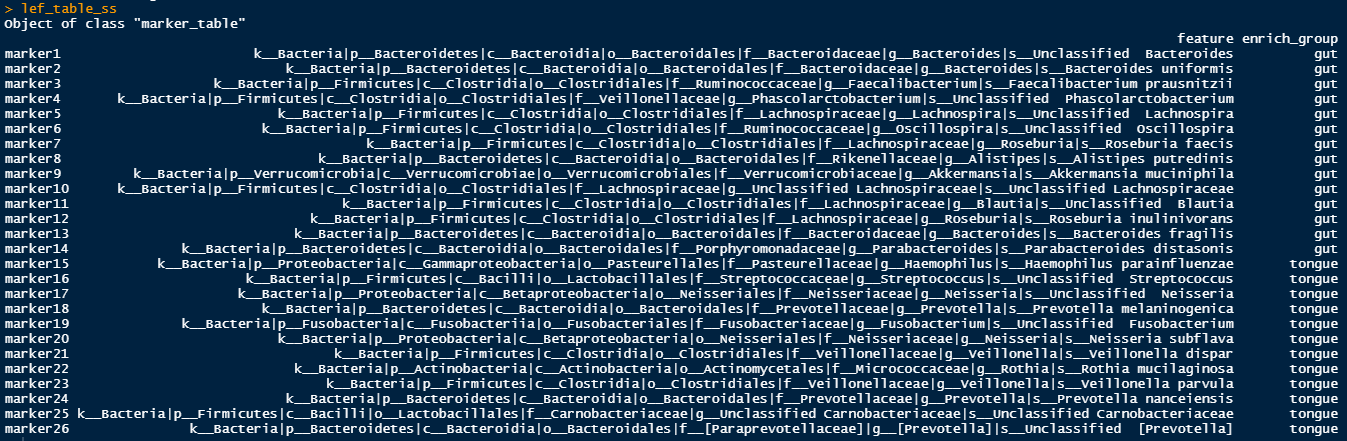
끄읏
반응형
