작성일 : 2022.10.20
if (!requireNamespace("BiocManager", quietly = TRUE)) {
install.packages("BiocManager")
}
BiocManager::install("microbiomeMarker")이걸로 처음에 다운받고
공식 튜토리얼(https://www.bioconductor.org/packages/devel/bioc/vignettes/microbiomeMarker/inst/doc/microbiomeMarker-vignette.html) 을 하려고 보니
library(microbiomeMarker) #
data(kostic_crc)
kostic_crc
mm_test <- run_lefse( kostic_crc,
wilcoxon_cutoff = 0.01,
norm = "CPM",
group = "DIAGNOSIS",
kw_cutoff = 0.01,
multigrp_strat = TRUE,
lda_cutoff = 4
)여기서부터 오류가 뜨면서 안된다
그래서 1. microbiomeMarker 패키지를 삭제하고 다시 깔아보았더니 아래와 같은 메세지가 뜬다
'rlang’ 1.0.5 is already loaded but rlang >= 1.0.6 is required
sessionInfo() 이걸로 확인해보니 1.0.5버전으로 되어있다
어떤분은 백신 프로그램때문이라는 사람도 있고, install.packeges("rlang")만 하면 된다는 사람도 있다.
나는 install.packeges("rlang") 이걸 해봐도 계속 1.0.5버전으로 깔렸다.
그래서 2. rlang을 삭제 후 다시 깔았더니 1.0.6으로 잘 깔렸다.
install_version("rlang", version = "1.0.6")이후 튜토리얼을 진행해 보았다.
data(kostic_crc)
kostic_crc
mm_test <- run_lefse( kostic_crc,
wilcoxon_cutoff = 0.01,
norm = "CPM",
group = "DIAGNOSIS",
kw_cutoff = 0.01,
multigrp_strat = TRUE,
lda_cutoff = 4
)
plot_ef_bar(mm_test)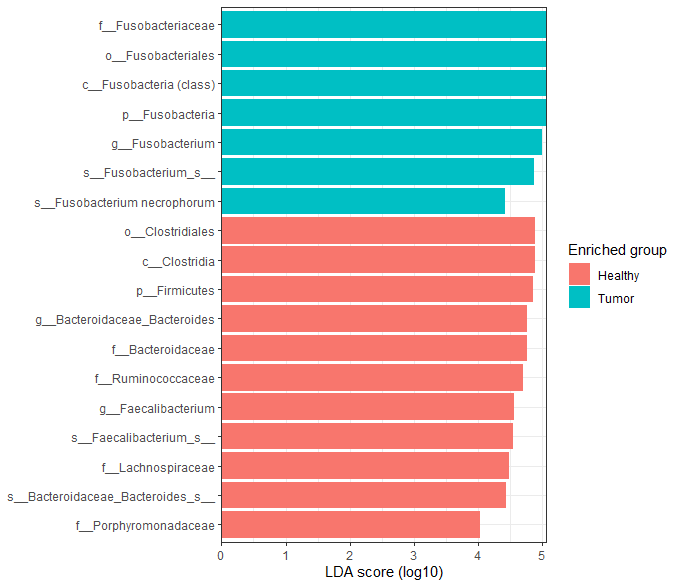
결과가 아주 잘 나온다 ^0^
이제 이 튜토리얼을 활용하여 moving-picture data에 한번 도입해본 후, 교수님이 주신 샘플에도 이후에 적용해봐야겠다
반응형
