
각 Phylum이 몇 퍼센트를 차지하는지 계산해 보자. 데이터는 Phyloseq의 기본 데이터 GlobalPatterns을 사용한다.
| 데이터 불러오기
library(phyloseq)
library(dplyr)
data("GlobalPatterns")
GlobalPatterns
# read count table -> relative abundance table
rel <- transform_sample_counts(GlobalPatterns, function (x) x/sum(x))
# melt
melt <- rel %>%
tax_glom(taxrank = "Phylum") %>% # 데이터의 크기를 줄이기 위해 Phylum 레벨로 리드 수를 합한다
psmelt()
준비된 melt 데이터를 살펴보자.
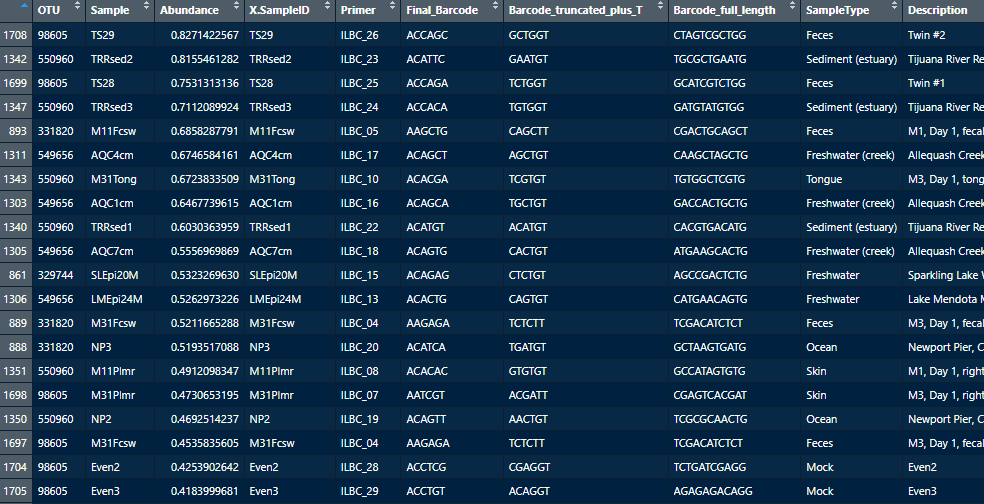
각각의 OTU, taxonomy, metadata가 따로 있는 biom format의 파일에서, 각 OTU가 어떤 샘플에서 어느 정도의 Abundance를 가지는지 데이터의 구성이 바뀌었다. 위 데이터를 이용하여 전체 샘플에 어떤 Phylum이 몇 %를 차지하는지 알아보자.
| Phylum % 계산하기
melt %>%
count(Phylum, wt = Abundance, name = "Abundance") %>%
mutate(percentage = 100 * Abundance / sum(Abundance)) %>%
head여기서 count는 각 Phylum의 abundance를 계산하고, mutate는 각 Phylum의 %를 계산하여 새로운 데이터를 만든다.
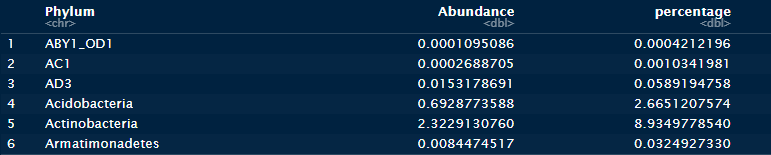
각 Phylum별로 Abundance의 합과, 각 Phylum이 차지하는 퍼센트를 Top 6만 뽑아서 관찰하였다.
하지만 percentage별로 정렬해서 다시 보고 싶다면 arrange함수를 이용하자.
melt %>%
count(Phylum, wt = Abundance, name = "Abundance") %>%
mutate(percentage = 100 * Abundance / sum(Abundance)) %>%
arrange(-percentage) %>%
head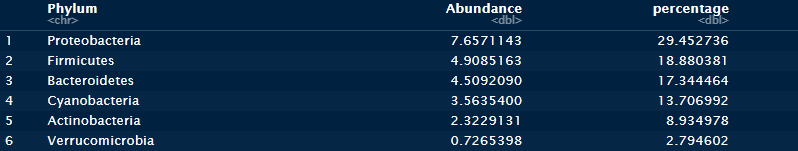
| 샘플 타입에 따른 Phylum abundance 평균 값 계산하기
일단 예제 데이터의 샘플 데이터를 살펴보자
sample_data(GlobalPatterns) %>% View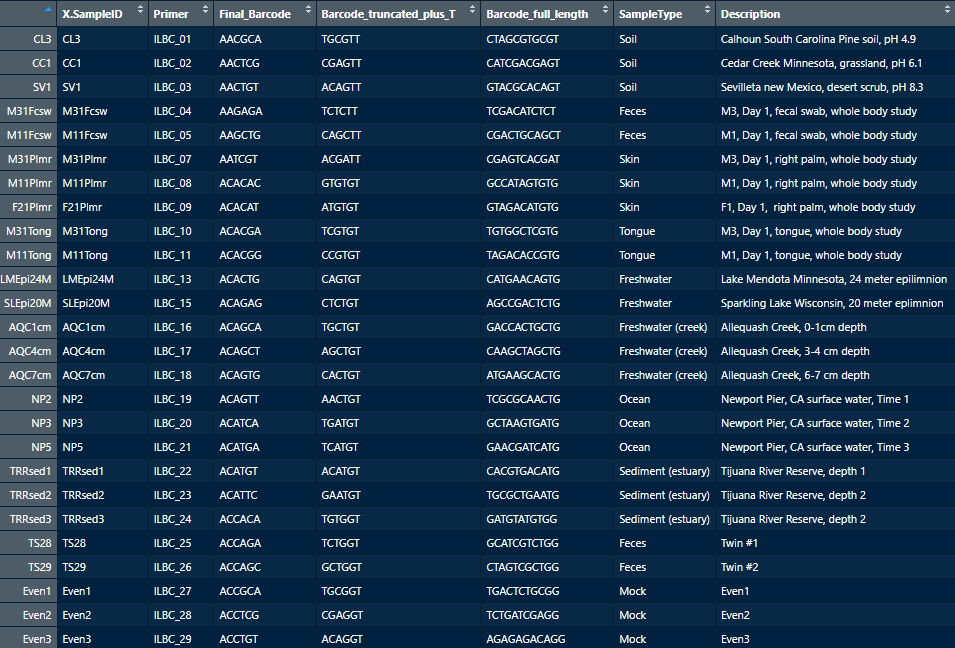
여기서 SampleType 열을 보면, 각 샘플이 샘플링된 지역을 표시하고있다. 우리는 이 타입에 따른 각 Phylum의 풍부도를 관찰해 보자. 이때 한 영역을 묶어서 계산하는 것은 함수 group_by()를 이용한다.
계산하기 전에 각 타입별로 Phylum의 종류가 많음으로 샘플의 0.01 이상의 abundance를 보이는 taxa만 추출해서 계산해보자
GlobalPatterns
rel <- transform_sample_counts(GlobalPatterns, function (x) x/sum(x))
rel.01 = filter_taxa(rel, function(x) sum(x) > .01, TRUE)
melt <- rel.01 %>% tax_glom(taxrank = "Phylum") %>% psmelt()
- 평균 abundance계산
type_abundance <- melt %>%
count(SampleType,Phylum, wt = Abundance, name = "Abundance") %>%
group_by(SampleType, Phylum) %>%
summarise(mean_abundance = mean(Abundance)) %>%
ungroup()
- Sample type에 따른 Phylum의 평균 abundance 값을 그림으로 나타내기
ggplot(type_abundance, aes(x = Phylum, y = mean_abundance,
label = str_c(mean_abundance, "%"))) +
geom_col() +
facet_wrap(~ SampleType) +
labs(title = "Mean abundance of each phylum at each type",
x = "SampleType",
y = "Mean relative abundance (%)")+
theme(axis.text.x.bottom = element_text(angle = 90, vjust = 0.4, hjust=1))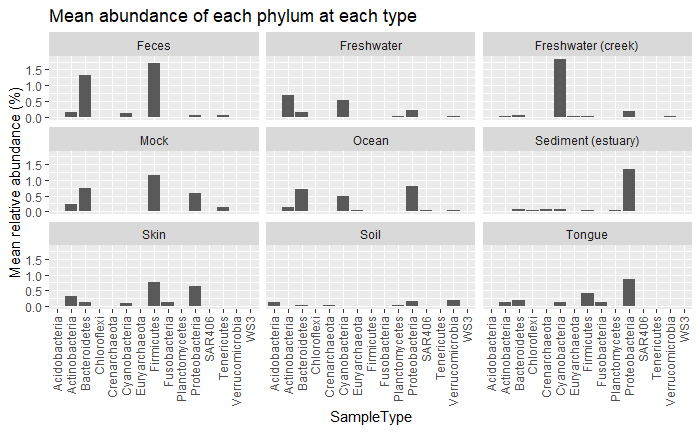
| 출처
- Wilkins, D., Tong, X., Leung, M.H.Y. et al. Diurnal variation in the human skin microbiome affects accuracy of forensic microbiome matching. Microbiome 9, 129 (2021). https://doi.org/10.1186/s40168-021-01082-1
- 위 논문의 코드 : https://github.com/wilkox/diurnal_variation
